筑波大学生命科学動物資源センター 久野朗広先生、水野聖哉先生インタビュー

KOマウス作製のためのCRISPR/Cas9デザインを全自動でできるウェブツール「KOnezumi」の開発
近年、CRISPRをはじめとするゲノム編集技術の急速な進歩により、ノックアウトマウスを作製するために必要な実験にかかる時間とコストは劇的に減少した。
しかし、実験に移る前に必要な、遺伝子の切断部位の設計などといった、具体的な遺伝学的情報を取得することは依然として多くの労力を費やす必要がある。
筑波大学生命科学動物資源センター 久野朗広氏、水野聖哉氏は、このマウス遺伝学情報を統合的に取得するウェブツールを開発され、これによって利用者は、ノックアウトしたいマウスの遺伝子名をツールに入力するだけで、CRISPR/Cas9システムにおけるguide RNA(以下gRNA)の標的配列を自動的に取得することが出来るという。
今回、この画期的なウェブツールの詳細についてお二人にお話を伺った。
久野:筑波大学動物資源センターではノックアウトマウス作製のために必要な情報を全自動で入手できるWebサービス、KOnezumiを公開しました。KOnezumiの利点として特に強調したいところは2つあります。
第一に「使い方が簡単」であることです。利用者の方は興味のある遺伝子名を入力するだけで、あとの作業はすべてKOnezumiが行います。
ゲノムブラウザから配列を入手して、CRISPR gRNA設計ツールにかけ、Primerの設計をして…といった手順を人が実行する必要がなく、簡単かつ速やかにノックアウトのデザインを完了できるということがKOnezumiを使う一番の利点です。
もう一つの利点は「信頼性の高いノックアウト戦略を提供する」ことです。
遺伝子ノックアウトの考え方にもいろいろとあり、例えば標的エクソン内を1箇所切断することでindel変異を導入する方法があります。
こちらは切断効率が良くて広く使われる方法ですが、indel変異はランダムに起こるものであることから実際に遺伝子機能が欠損しているのかはっきりしない点がデメリットです。
特にノックアウトマウスの作製では効率よりも目的の遺伝子の機能を確実に欠損することが優先されるため、KOnezumiは標的エクソンの両端2箇所を切断して取り除く方法を採用しました。
そこで「どこのエクソンを標的とするか」が重要になりますが、国際マウス表現型解析コンソーシアム(IMPC)/国際ノックアウトマウスコンソーシアム(IKMC)が、
「すべての転写物バリアントに含まれること、かつそのエクソンの欠損により(フレームシフト変異が起こり)コーディング領域の半分以上が異なる配列となるエクソンを標的とする」
との方針でマウスの全遺伝子を網羅的にノックアウトするプロジェクトを遂行しており、彼らが標的としたエクソンの情報は一般に公開されています。
KOnezumiはその公開情報をもとに標的エクソンを選択しており、任意のエクソンを削るよりもより信頼性の高いノックアウトを実現することができると考えています。
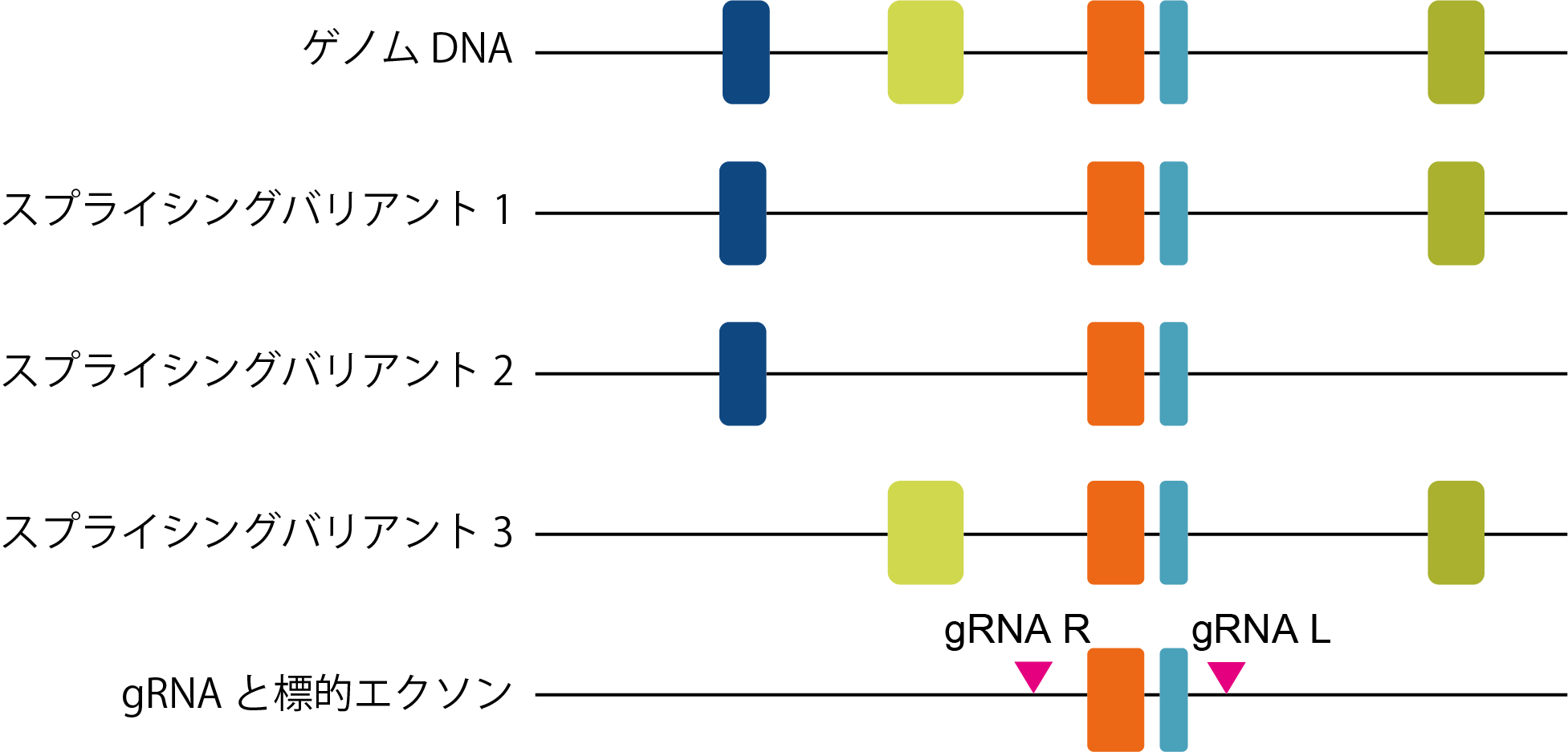
KOnezumiでは、全ての転写物バリアントに含まれ、かつそのエクソンの欠損により(フレームシフトが起こり)コーディング領域の半分以上が異なる配列となるエクソンを標的としています。
久野:KOnezumiの使い方はとても簡単です。
興味のある遺伝子名(複数入力可能)を入力していただくだけです。
あとは全自動で結果を出力します。
もともと複雑な手順をなくしたいというモチベーションで開発しましたので、KOnezumiはどなたでも簡単に使えることを最重要に考えています。
レポート結果は大きく分けて5種類あり、(1) 遺伝子情報・ノックアウト戦略の概要について、(2) gRNAの候補一覧、(3) ジェノタイピングPCRプライマーの候補一覧、(4) 切断されるエクソンの配列、(5) 切断後のmRNA配列、を表示いたします。
特に(3) ジェノタイピングPCRプライマーの候補一覧と(5) 切断後のmRNA配列の情報はエクソンの両端を切除するノックアウト方法で活きてくる情報です。
ジェノタイピングPCRプライマーは電気泳動のバンドサイズでホモとヘテロをわけられるように3種類のプライマー情報を提供します。
また、切断後のmRNA配列を見ることでそのmRNAがナンセンス変異依存mRNA分解機構(NMD)によって破壊される可能性を考えることができます。
これらの情報から、高い確実性を持ってノックアウトマウスの遺伝型を同定することができます。
現状(2019 年 3 月時点)では約1万2000遺伝子に対応しており、これからも対応する遺伝子数を増やしていく予定です。
もし興味のある遺伝子が対応していなくても、適当な遺伝子でKOnezumiのレポートを眺めていただき、ノックアウトデザインを考える参考になれば嬉しく思います。
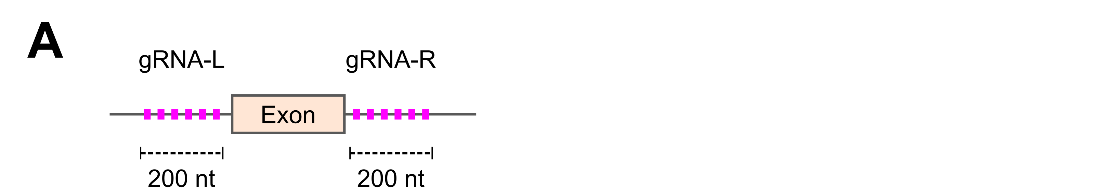
(A) 標的となるエクソンの両側200nt以内にgRNAをデザインします(■:gRNA)。
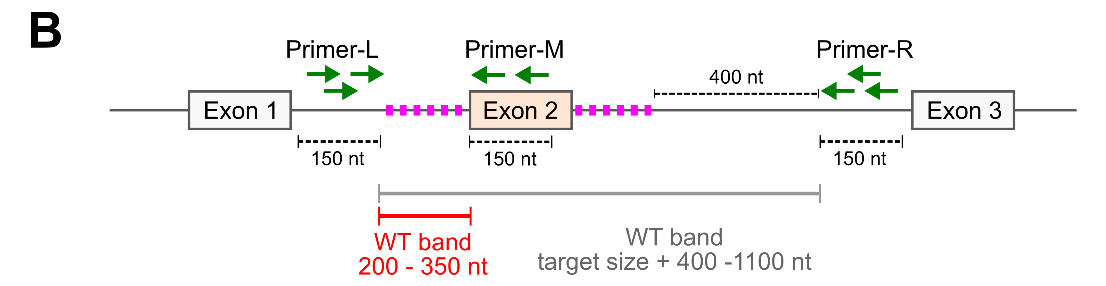
(B)デザインしたgRNAの外側にPrimer-LとRを、欠損予定箇所にPrimer-Mをデザインします。WT bandは、PCRを行った際のPCR産物のサイズを示しています。
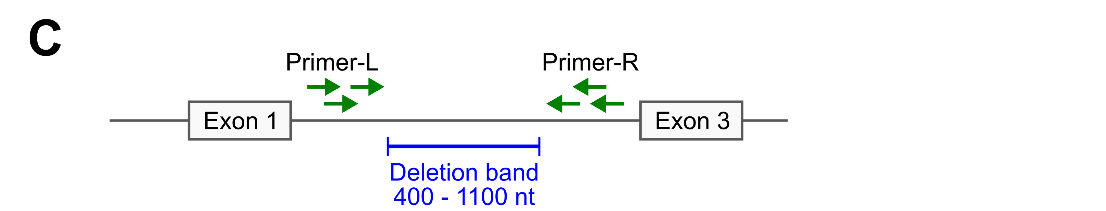
(C) Deletion bandは、標的エクソンがノックアウトされた際のPCR産物のサイズを示しています。
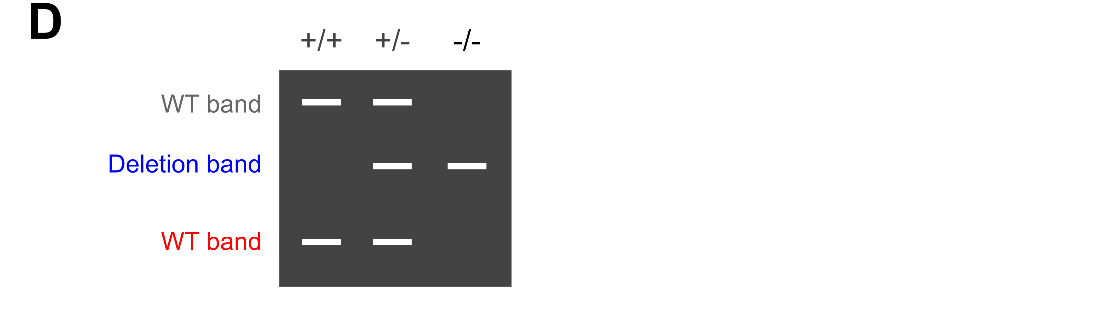
(D) +/+ :IMPC/IKMC方針規格のノックアウトができていない場合
+/- :ヘテロでノックアウトがされている状態
-/- :ノックアウトホモの状態
(図A~D:http://dx.doi.org/10.1093/bioinformatics/btz090 の Supplementary dataを改変)
KOnezumiでは、上記の方針に基づいたgRNA、プライマーデザインを提供するだけでなく、ターゲットエクソンの配列や、Deletion 後の配列も提供しています。
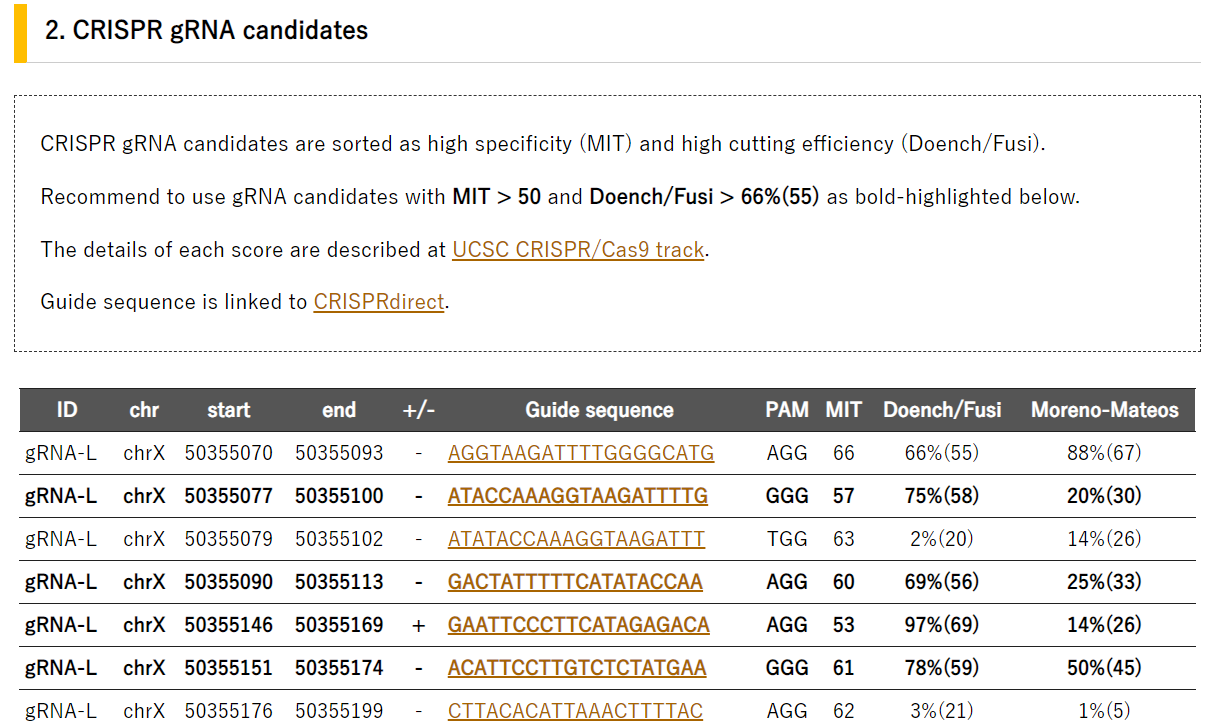
KOnezumiでHPRT遺伝子を検索した際のgRNA設計データの一部を示しています。遺伝子名を入力するだけですぐに結果が示されます。
» KOnezumiの使用方法と得られるレポートの詳細はこちら
久野:KOnezumiはおよそ2年前に開発を始めました。
当時の私はWETとDRYの両方に取り組んでおり、DRYで絞り込んだある遺伝子のノックアウト細胞を樹立したいと考えて遺伝子改変のプロである水野先生に相談いたしました。
水野先生にはひとつひとつ大変丁寧にご指導いただきました。
このときに教えていただいた内容がほとんどそのままKOnezumiのレポート結果になっています。
しかしながら私のような初心者では複数のステップのどこかで間違えたり、そもそもやり方を忘れてしまう可能性もあったりと、はたして自分にできるのか全く自信がありませんでした。
水野先生に全部お任せすることも頭をよぎりましたが、幸いなことに水野先生が使用したツールの大半はデータが公開されていたり、APIを提供していたりとプログラムに落とし込むことができそうだったので、実験ノート代わりのつもりでプログラムを書くことにしました。
1ヶ月ほどである程度一連の流れをプログラム化することができ、内心では満足したのでラボミーティングで発表したのですが、反応は良くありませんでした。
当初は黒い画面(ターミナル)でしか動かなかったので、利用するには敷居が高かったからです。
ここから自分だけのものではなく、広く皆さまに使っていただくツールを開発することが目的となりました。
以降は使いやすさを求めて試行錯誤を経て(ターミナル→R Shinyアプリ→Webツール)、最終的に動物資源センターのご協力を得てWebツールとして公開することができました。
開発途中で水野先生から「切断後のmRNAのPTC(未成熟終止コドン)の位置とかも表示できたら嬉しいです。できれば色付きで!」を筆頭とするムチャ振りをいただきそのたびに頭を抱えましたが、大変重要なフィードバックをいただき、これらはすべてKOnezumiの強みになっています。
まだ実現できていないご指摘も多数ありますので、引き続き開発していきます!
水野:久野先生が開発されたKOnezumiはゲノム情報のみならず、各種有用なデータベースへのリンクが素早く表示されますので、既にgRNAのデザインに精通している研究者にも使い勝手がよいツールです。
私は、まずは従来通りに自分でgRNAの設計を実施し、その後にKOnezumiのレポートと自分のデザインを比較することで、二重チェックを行っています。
また、編集後のアレルから発現することが予測されるmRNA(cDNA)配列が表示されるのも大変有難い機能だと感じています。
それは、上記の様なNMDの予測のみならず、RT-PCRのPrimerを設計する際にも使用できるためです。
以上の様に、初心者の方から熟練者の方まで色々なKOnezumiの使用法があると思いますので、まずは一度試してみて欲しいです。
謝辞
久野:本研究をサポートしてくださいました筑波大学動物資源センター 水野先生、高橋智教授にお礼申し上げます。
また執筆の機会を与えていただきましたIDT 藤森様、安井様に感謝いたします。
しかし、実験に移る前に必要な、遺伝子の切断部位の設計などといった、具体的な遺伝学的情報を取得することは依然として多くの労力を費やす必要がある。
筑波大学生命科学動物資源センター 久野朗広氏、水野聖哉氏は、このマウス遺伝学情報を統合的に取得するウェブツールを開発され、これによって利用者は、ノックアウトしたいマウスの遺伝子名をツールに入力するだけで、CRISPR/Cas9システムにおけるguide RNA(以下gRNA)の標的配列を自動的に取得することが出来るという。
今回、この画期的なウェブツールの詳細についてお二人にお話を伺った。
KOnezumiで提供したいKOデザインとは - 簡便、かつ信頼性の高いWebツール
久野:筑波大学動物資源センターではノックアウトマウス作製のために必要な情報を全自動で入手できるWebサービス、KOnezumiを公開しました。KOnezumiの利点として特に強調したいところは2つあります。
第一に「使い方が簡単」であることです。利用者の方は興味のある遺伝子名を入力するだけで、あとの作業はすべてKOnezumiが行います。
ゲノムブラウザから配列を入手して、CRISPR gRNA設計ツールにかけ、Primerの設計をして…といった手順を人が実行する必要がなく、簡単かつ速やかにノックアウトのデザインを完了できるということがKOnezumiを使う一番の利点です。
もう一つの利点は「信頼性の高いノックアウト戦略を提供する」ことです。
遺伝子ノックアウトの考え方にもいろいろとあり、例えば標的エクソン内を1箇所切断することでindel変異を導入する方法があります。
こちらは切断効率が良くて広く使われる方法ですが、indel変異はランダムに起こるものであることから実際に遺伝子機能が欠損しているのかはっきりしない点がデメリットです。
特にノックアウトマウスの作製では効率よりも目的の遺伝子の機能を確実に欠損することが優先されるため、KOnezumiは標的エクソンの両端2箇所を切断して取り除く方法を採用しました。
そこで「どこのエクソンを標的とするか」が重要になりますが、国際マウス表現型解析コンソーシアム(IMPC)/国際ノックアウトマウスコンソーシアム(IKMC)が、
「すべての転写物バリアントに含まれること、かつそのエクソンの欠損により(フレームシフト変異が起こり)コーディング領域の半分以上が異なる配列となるエクソンを標的とする」
との方針でマウスの全遺伝子を網羅的にノックアウトするプロジェクトを遂行しており、彼らが標的としたエクソンの情報は一般に公開されています。
KOnezumiはその公開情報をもとに標的エクソンを選択しており、任意のエクソンを削るよりもより信頼性の高いノックアウトを実現することができると考えています。
IMPC/IKMCのノックアウト方針とKOnezumiの設計の概念図

KOnezumiでは、全ての転写物バリアントに含まれ、かつそのエクソンの欠損により(フレームシフトが起こり)コーディング領域の半分以上が異なる配列となるエクソンを標的としています。
KOnezumiは遺伝子名を入力するだけですぐに結果が出る
久野:KOnezumiの使い方はとても簡単です。
興味のある遺伝子名(複数入力可能)を入力していただくだけです。
あとは全自動で結果を出力します。
もともと複雑な手順をなくしたいというモチベーションで開発しましたので、KOnezumiはどなたでも簡単に使えることを最重要に考えています。
レポート結果は大きく分けて5種類あり、(1) 遺伝子情報・ノックアウト戦略の概要について、(2) gRNAの候補一覧、(3) ジェノタイピングPCRプライマーの候補一覧、(4) 切断されるエクソンの配列、(5) 切断後のmRNA配列、を表示いたします。
特に(3) ジェノタイピングPCRプライマーの候補一覧と(5) 切断後のmRNA配列の情報はエクソンの両端を切除するノックアウト方法で活きてくる情報です。
ジェノタイピングPCRプライマーは電気泳動のバンドサイズでホモとヘテロをわけられるように3種類のプライマー情報を提供します。
また、切断後のmRNA配列を見ることでそのmRNAがナンセンス変異依存mRNA分解機構(NMD)によって破壊される可能性を考えることができます。
これらの情報から、高い確実性を持ってノックアウトマウスの遺伝型を同定することができます。
現状(2019 年 3 月時点)では約1万2000遺伝子に対応しており、これからも対応する遺伝子数を増やしていく予定です。
もし興味のある遺伝子が対応していなくても、適当な遺伝子でKOnezumiのレポートを眺めていただき、ノックアウトデザインを考える参考になれば嬉しく思います。
KOnezumiにおけるgRNAおよびジェノタイピングPCRプライマーのデザイン
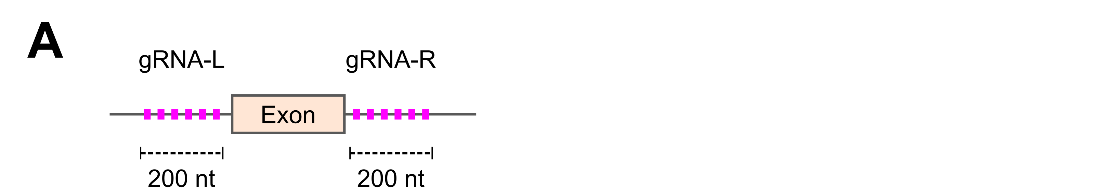
(A) 標的となるエクソンの両側200nt以内にgRNAをデザインします(■:gRNA)。

(B)デザインしたgRNAの外側にPrimer-LとRを、欠損予定箇所にPrimer-Mをデザインします。WT bandは、PCRを行った際のPCR産物のサイズを示しています。

(C) Deletion bandは、標的エクソンがノックアウトされた際のPCR産物のサイズを示しています。

(D) +/+ :IMPC/IKMC方針規格のノックアウトができていない場合
+/- :ヘテロでノックアウトがされている状態
-/- :ノックアウトホモの状態
(図A~D:http://dx.doi.org/10.1093/bioinformatics/btz090 の Supplementary dataを改変)
KOnezumiでは、上記の方針に基づいたgRNA、プライマーデザインを提供するだけでなく、ターゲットエクソンの配列や、Deletion 後の配列も提供しています。
KOnezumiから得られるレポート

KOnezumiでHPRT遺伝子を検索した際のgRNA設計データの一部を示しています。遺伝子名を入力するだけですぐに結果が示されます。
» KOnezumiの使用方法と得られるレポートの詳細はこちら
2年の歳月を経て、WETをDRYに落とし込んだ
久野:KOnezumiはおよそ2年前に開発を始めました。
当時の私はWETとDRYの両方に取り組んでおり、DRYで絞り込んだある遺伝子のノックアウト細胞を樹立したいと考えて遺伝子改変のプロである水野先生に相談いたしました。
水野先生にはひとつひとつ大変丁寧にご指導いただきました。
このときに教えていただいた内容がほとんどそのままKOnezumiのレポート結果になっています。
しかしながら私のような初心者では複数のステップのどこかで間違えたり、そもそもやり方を忘れてしまう可能性もあったりと、はたして自分にできるのか全く自信がありませんでした。
水野先生に全部お任せすることも頭をよぎりましたが、幸いなことに水野先生が使用したツールの大半はデータが公開されていたり、APIを提供していたりとプログラムに落とし込むことができそうだったので、実験ノート代わりのつもりでプログラムを書くことにしました。
1ヶ月ほどである程度一連の流れをプログラム化することができ、内心では満足したのでラボミーティングで発表したのですが、反応は良くありませんでした。
当初は黒い画面(ターミナル)でしか動かなかったので、利用するには敷居が高かったからです。
ここから自分だけのものではなく、広く皆さまに使っていただくツールを開発することが目的となりました。
以降は使いやすさを求めて試行錯誤を経て(ターミナル→R Shinyアプリ→Webツール)、最終的に動物資源センターのご協力を得てWebツールとして公開することができました。
開発途中で水野先生から「切断後のmRNAのPTC(未成熟終止コドン)の位置とかも表示できたら嬉しいです。できれば色付きで!」を筆頭とするムチャ振りをいただきそのたびに頭を抱えましたが、大変重要なフィードバックをいただき、これらはすべてKOnezumiの強みになっています。
まだ実現できていないご指摘も多数ありますので、引き続き開発していきます!
初心者から上級者まで、多くの研究者に有用なツール
水野:久野先生が開発されたKOnezumiはゲノム情報のみならず、各種有用なデータベースへのリンクが素早く表示されますので、既にgRNAのデザインに精通している研究者にも使い勝手がよいツールです。
私は、まずは従来通りに自分でgRNAの設計を実施し、その後にKOnezumiのレポートと自分のデザインを比較することで、二重チェックを行っています。
また、編集後のアレルから発現することが予測されるmRNA(cDNA)配列が表示されるのも大変有難い機能だと感じています。
それは、上記の様なNMDの予測のみならず、RT-PCRのPrimerを設計する際にも使用できるためです。
以上の様に、初心者の方から熟練者の方まで色々なKOnezumiの使用法があると思いますので、まずは一度試してみて欲しいです。
謝辞
久野:本研究をサポートしてくださいました筑波大学動物資源センター 水野先生、高橋智教授にお礼申し上げます。
また執筆の機会を与えていただきましたIDT 藤森様、安井様に感謝いたします。
参考文献等
Kuno A, Mizuno S, Takahashi S. Bioinformatics 2019
http://dx.doi.org/10.1093/bioinformatics/btz090
KOnezumiリンク
http://www.md.tsukuba.ac.jp/LabAnimalResCNT/KOanimals/konezumi.html
筑波大学生命科学動物資源センターが実施する遺伝子改変マウス作製受託事業
http://www.md.tsukuba.ac.jp/LabAnimalResCNT/sakusei.html
http://dx.doi.org/10.1093/bioinformatics/btz090
KOnezumiリンク
http://www.md.tsukuba.ac.jp/LabAnimalResCNT/KOanimals/konezumi.html
筑波大学生命科学動物資源センターが実施する遺伝子改変マウス作製受託事業
http://www.md.tsukuba.ac.jp/LabAnimalResCNT/sakusei.html
研究者紹介
 |
久野 朗広
(くの あきひろ) MD, Ph.D
|
 |
水野 聖哉
(みずの せいや) Ph.D
|
製品フォーカス
Alt-R® CRISPR-Cas9 System
Alt-R® CRISPR-Cas9 Systemには、ゲノム編集を行うためのキーとなる試薬が揃っています。S. pyogenesの持つCRISPR-Cas9 systemに由来する本システムは、下記の様に多数のアドバンテージがあります。- 他社の手法と比較し、オンターゲット率が向上します。
- リポフェクションやエレクトロポレーションで、効率よくRNPが導入できます。
- sgRNAやCas9 mRNAを導入した際に見られる、自然免疫を惹起しません。
Alt-R® CRISPR-Cas12a System
Alt-R® CRISPR-Cas12a Systemを用いると、これまでCRISPR-Cas9で切断できなかったサイトでゲノム編集を行えます。Cpf1でDNAを切断すると、5'突出末端となります。本試薬には下記の特徴があります。
- AT-richなゲノム領域でもゲノム編集が行えます。
- Cas9では切断できなかったサイトを補填できます。
- Cas12a NucleaseとcrRNAのみで、ゲノム編集を行えます。tracrRNAは必要ありません。
- エレクトロポレーション法で効率よくRNPを導入できます。