理化学研究所バイオリソース研究センター 綾部信哉先生インタビュー
CRISPR/Cas9, Cas12aシステムによるゲノム編集マウスの作製
~ ノックアウトからノックインへ ~
2021年までに、マウスの全ての遺伝子機能を解析する事を目的としたプロジェクト「国際マウス表現型解析コンソーシアム(IMPC)」が日本を含む15の国・地域の国際連携により進められている。
このIMPC国内拠点である理研バイオリソース研究センター実験動物開発室の綾部信哉氏は、プロジェクトにおけるノックアウトマウスのみならず、ノックインマウスも多く作出しているという。
今回、この壮大なプロジェクトの展望と、ノックアウト・ノックインマウス作出の詳細についてお話を伺った。
IMPCのプロジェクトから発展した、CRISPRによるノックインマウスの作製
理研バイオリソース研究センター 動物実験開発室では、社会・研究ニーズに応える最先端のモデルマウスを収集・保存・品質管理・提供している。
その中で、IMPCへの参画により、マウスゲノムに存在するたんぱく質をコードする全ての遺伝子についてのノックアウトマウスを作製し、各ノックアウトマウスの表現系データと併せたデータベースとして研究コミュニティに提供している。
各ノックアウトマウスの作製には、もちろんCRISPR/Cas9システムが用いられている。
綾部氏は化学合成されたgRNAと、Cas9ヌクレアーゼタンパク質の複合体(RNP)を、主にエレクトロポレーションにより受精卵に導入する方法でノックアウトマウスを作製している。
近日はこれに加えて、多くのノックインマウスの作製にも取り組んでいるという。
「もともとはノックアウトマウスを作製することを目的としたプロジェクトでした。
2012年から始まったプロジェクトですが、途中からCRISPR/Cas9というテクノロジーが登場したため、より簡便にノックアウトマウスを作製することが出来るようになりました。
そこで、このプロジェクトをどのように発展させていくかという事を考えた際に、疾患の原因となるポイントミューテーションを入れたマウスや、loxP配列を挿入したコンディショナルノックアウトマウスを作製することで、コミュニティに更なる貢献が出来ると考えました。
こちらはまだIMPCのオフィシャルな活動ではなく、パイロット運用なのですが、ポイントミューテーションを入れたマウスをIMPCのプラットフォームに乗せて解析することにより、ヒト疾患モデルとしてのマウスの価値をさらに高める事ができるはずだと考えます。
また、コンディショナルノックアウトマウス作製においてはexonの両端のintronを一度に2箇所カットする際に、loxP配列のノックインを試みることで、ノックインされたマウスと、エクソンが落ちたノックアウトマウスの両方を取得できて一石二鳥です。」
実験手法についてもIMPCに加盟している機関で行われる月1回のミーティングで共有されているという。
「受精卵に対してエレクトロポレーションを行うという手法は、いち早く日本から発信する形となりました。インジェクションは技術的にそもそも難しいため、技術者の育成も困難です。
現在、ノックアウトやポイントミューテーション、タグの挿入くらいであれば、全てエレクトロポレーションで実施しています。
また、ノックインにはIDTのUltramer PAGE精製がいいよという話が参画機関の研究員からあり、私も当初はそれを使っておりましたが、現在は脱塩でもうまくいくので、脱塩に切り替えました(笑)」
Cas12aによるノックアウト・ノックインマウスの作成
現在、異なる特徴を持つCRISPR-Cas酵素が次々と発見されている。
中でもCas9と同様に、ゲノム編集に利用されているCas12a(Cpf1)という酵素がある。
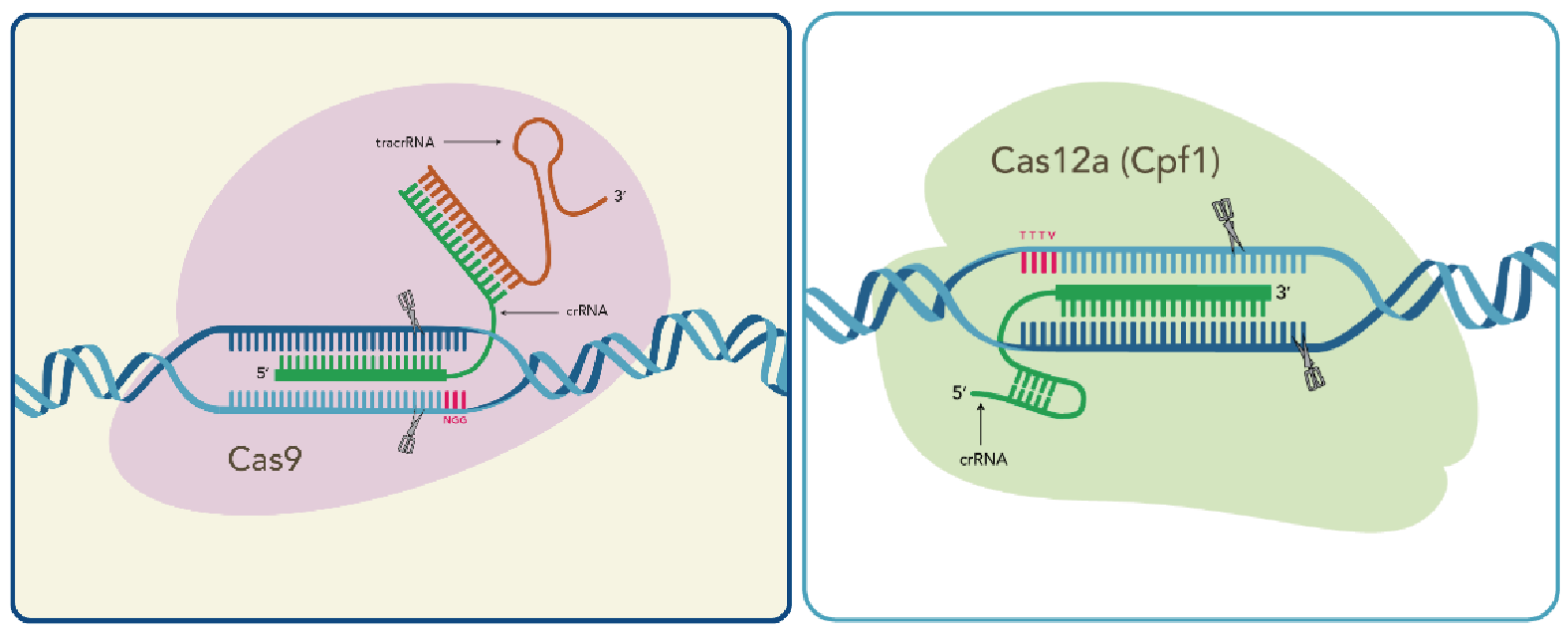
Cas9とCas12aはどちらもgRNAと複合体を形成して標的となる2本鎖DNAを切断する活性を持つが、両者には違いもある。Cas9はDNAを垂直にカットし、2本鎖がそろった平滑末端をつくるが、Cas12aは5’突出末端をつくる。
また、Cas9はNGGというPAMサイトを目印にDNAを切断するのに対し、Cas12aはTTTV(V : A, G, C)を目印にするので、異なる部位を切断することができる。
綾部氏はノックアウトだけではなく、ノックインにもCas12aを使用している。
「ノックアウトのみならず、ノックインにおいて、特にポイントミューテーションを入れる際にCas12aが有用なケースがあります。
ポイントミューテーションの導入においては、入れたいミューテーションに近い位置にNucleaseのCutサイトが来るようにgRNAを選択することが望ましいため、ミューテーションの近くにCas12aのPAMサイトがある場合はこちらを選択する事も考えられます。
CRISPR-Cas12aの標的への結合は、非特異的に周りの一本鎖DNAを分解する活性持っていることが知られています。
つまり、ドナーのssODNも切断してしまう可能性が高いと考えておりましたが、実際に行った実験では、目的の変異体が取れてきているため、ノックイン実験への使用も不可能では無いということを示しました。」
実験プロトコル
世界中の研究者と随時情報共有を行い、ご自身でも新たな情報をアグレッシブに取得されている綾部氏。この度、実際に実施されているCas9, Cas12aを用いたノックインマウス作製方法の詳細、およびノウハウを公開頂いた。
■ 受精卵の種類: C57BL/6N系統
試薬量:1回のエレクトロポレーションあたり(5-mm電極の場合)50 μl程度、もしくは(1-mm 電極の場合)5 μl程度、Opti-MEM® I Reduced Serumで希釈
- crRNA:(Cas12aの場合)final 70 ng/μl Alt-R® CRISPR-Cas12a(Cpf1) crRNA
- tracrRNA:(Cas9の場合)gRNAとしてfinal 250 ng/μl、Alt-R® CRISPR-Cas9 crRNA, tracrRNA
- Cas9:final 400 ng/μl Alt-R™ S.p. Cas9 Nuclease V3
- Cas12a:final 400 ng/μl Alt-R™ A.s. Cas12a(Cpf1) Nuclease V3
- ドナーDNA:final 100 ng/μl Ultramer DNA(PAGE精製もしくは脱塩精製)
- エレクトロポレーター: NEPA21(ネッパジーン社製)
- オリゴの設計(ホモロジーアームの長さなど):60-80塩基のアームをつけています
- プロトコル:J Reprod Dev. 2019 Feb 8;65(1):1-5、をご参照ください
ノックインの際のコツ
■ gRNAやオリゴの設計について
- gRNAを設計する際には、CRISPORやKOnezumiなどを利用して、オフターゲットサイトの候補数が少ない配列を選んでいます。Cas12aを使う場合のオフターゲットサイト検索には例えばCas-OFFinderが利用できます。
- 特定の場所にノックインを行いたい場合はオフターゲットサイトの候補が多い配列をgRNAとして選ばなければならない場合がありますが、Cas9によるマウス受精卵ゲノム編集ではオフターゲットサイトはまれにしか編集されないため、ほとんど気にする必要は無いと考えています。詳細はプロトコルの論文をご参照ください。
- 特に点変異挿入時のオリゴについて、不思議なことに上記とは一致しませんが、ノックインされた領域をCas9が認識・切断して再編集されたと思われる事例を経験しているため、目的の点変異に加えてPAM配列やgRNAの3’側にサイレント変異を加えたオリゴを設計することで再切断を防いでいます。
- 胎生致死遺伝子や胎生致死となる変異を挿入する場合は、上記のサイレント変異のみを乗せたオリゴを併用してノックインを行ったり、そのオリゴだけを使った変異マウス作製を並行して進めておき次世代で片アレルのみの編集を行ったりしています。
■ エレクトロポレーションについて
- 凍結胚を使用する場合は、成熟メスに由来する受精卵を使用した方が個体復元成績が良い場合があります。
- ドナーのssDNAが複数ある場合は、合計が150 ng/µlを超えないようにすると復元率がよいです。
- 使用する培養液や室内環境、胚操作のプロトコルなどによって受精卵の状態はラボごとに若干異なるため、まずは既報などをもとに各ラボで最適な電気条件を検討することをお勧めします。
- エレクトロポレーションにはOpti-MEMを電極液として使用しています。gRNA complexを作ったのちにOpti-MEMで希釈することで、Duplex bufferの持ち込み量を最小限に抑えています。
- 使用するCasのモル濃度に対して1:1ではなく1:~5程度の濃い濃度でgRNAやcrRNAを使用することで、効率よく編集できる場合があります。
■ 特定の箇所で編集が起こらない場合
- 実験系が問題ないにもかかわらず編集が起こらない場合は、同一の配列でもsgRNAをgRNA complexに変えてみたり、Cas9ではなくCas12aを使ってみたりすることでうまくいく場合があります。
新たなツールが次々と開発されている分野なので、定期的に情報をアップデートして手法の幅を広げていただければと思います。
Reference
1. J Reprod Dev. 2019 Feb 8;65(1):1-5
 |
綾部 信哉(あやべ しんや)
|
製品フォーカス
Alt-R® CRISPR-Cas9 System
Alt-R® CRISPR-Cas9 Systemには、ゲノム編集を行うためのキーとなる試薬が揃っています。S. pyogenesの持つCRISPR-Cas9 systemに由来する本システムは、下記の様に多数のアドバンテージがあります。- 他社の手法と比較し、オンターゲット率が向上します。
- リポフェクションやエレクトロポレーションで、効率よくRNPが導入できます。
- sgRNAやCas9 mRNAを導入した際に見られる、自然免疫を惹起しません。
Alt-R® CRISPR-Cas12a System
Alt-R® CRISPR-Cas12a Systemを用いると、これまでCRISPR-Cas9で切断できなかったサイトでゲノム編集を行えます。Cpf1でDNAを切断すると、5'突出末端となります。本試薬には下記の特徴があります。
- AT-richなゲノム領域でもゲノム編集が行えます。
- Cas9では切断できなかったサイトを補填できます。
- Cas12a NucleaseとcrRNAのみで、ゲノム編集を行えます。tracrRNAは必要ありません。
- エレクトロポレーション法で効率よくRNPを導入できます。
Ultramer® DNA合成
Ultramer DNA合成では、45 ~ 200塩基の一本鎖DNAオリゴを合成できます。HDRドナーテンプレートとして多くの研究者様にお使いいただいております。詳しくはこちらをご参照ください。