KOnezumiのKOマウスの設計原理とgRNAとプライマーのデザイン、オーダー方法
筑波大学の久野先生が開発されたKOマウス作製のためのウェブツールであるKOnezumiに実際にHPRT遺伝子を入力し、得られたレポートを紹介しています。本レポートは2019年4月時点のレポートであり、今後の開発等によって異なる情報が排出される可能性があります。
KOnezumiからレポートを取得する
-
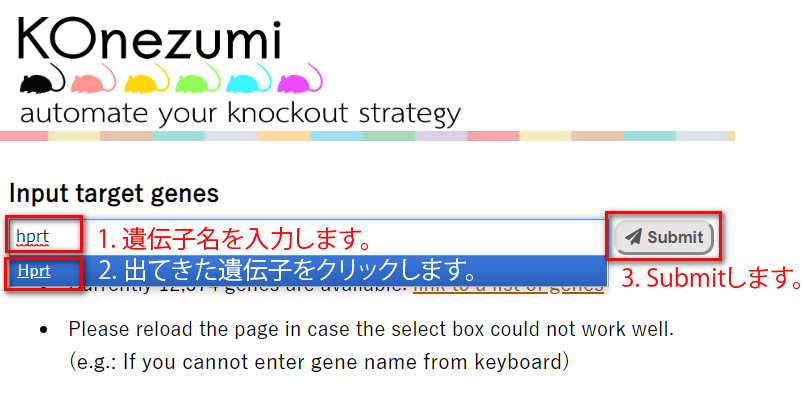
KOnezumiにアクセスします。

- 遺伝子名を入力し、出てきた遺伝子をクリックします。
今回は例としてHPRTで検索してみます。
入力が終わったら[Submit]をクリックします。
※クリックで拡大
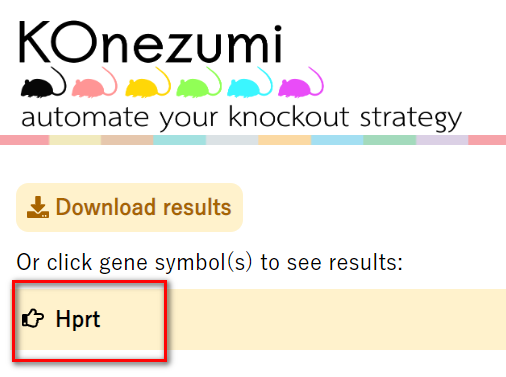
- もう1度HPRTをクリックします。(全画面で他種類の遺伝子を入力していると、ここで各遺伝子のレポートが表示されます。)
※クリックで拡大
- HPRTをクリックすると、5種のレポートについて見ることができます。
- 遺伝子情報・ノックアウト戦略の概要について
- gRNAの候補一覧
- ジェノタイピングPCRプライマーの候補一覧
- 切断されるエクソンの配列
- 切断後のmRNA配列
- 1. 遺伝子情報・ノックアウト戦略の概要について
KOnezumiの利点の一つは、「信頼性の高いノックアウト戦略を提供する」ことです。
KOnezumiでは、ノックアウトマウス作製のため国際マウス表現型解析コンソーシアム(IMPC)/国際ノックアウトマウスコンソーシアム(IKMC)の
「すべての転写物バリアントに含まれること、かつそのエクソンの欠損により(フレームシフト変異が起こり)コーディング領域の半分以上が異なる配列となるエクソンを標的とする」
という方針でノックアウトをデザインしています。
具体的にどのようにデザインするか、図を使って解説させていただきます。
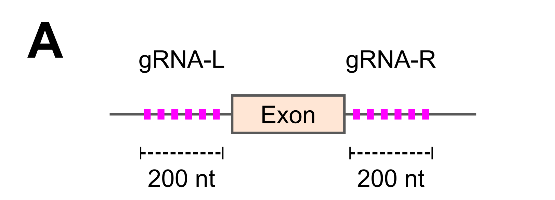
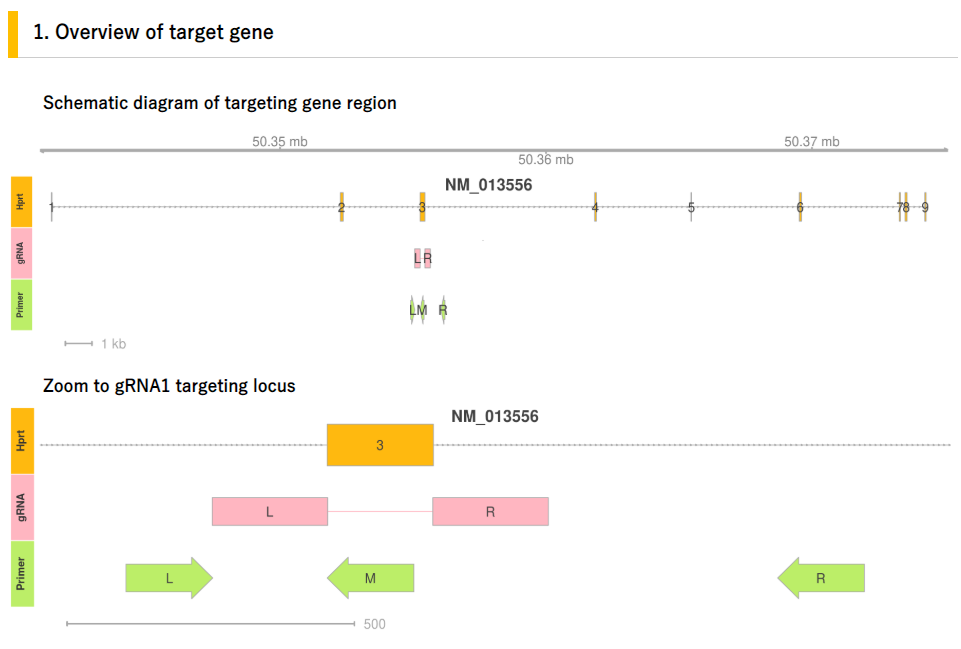
(A) 標的となるエクソンの両側200nt以内にgRNAをデザインします(■:gRNA)。
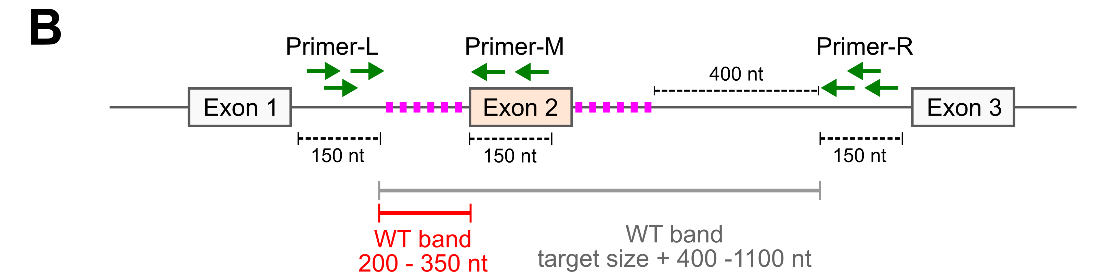
(B)デザインしたgRNAの外側にPrimer-LとRを、欠損予定箇所にPrimer-Mをデザインします。WT bandは、PCRを行った際のPCR産物のサイズを示しています。
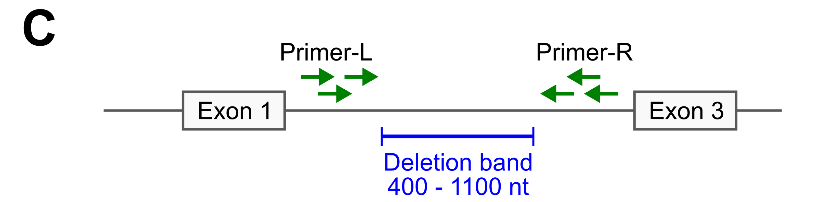
(C) Deletion bandは、標的エクソンがノックアウトされた際のPCR産物のサイズを示しています。
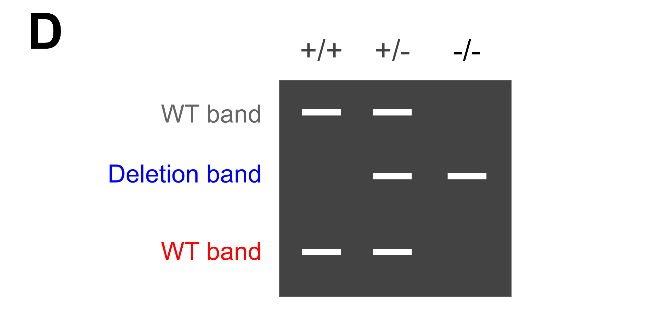
(D) +/+ :IMPC/IKMC方針のノックアウトができていない場合
+/- :ヘテロでノックアウトがされている状態
-/- :ノックアウトホモの状態
上記のうちHPRTに対する(A)および(B)の情報が下記の様に示されます。

※クリックで拡大 - 2. gRNAの候補一覧
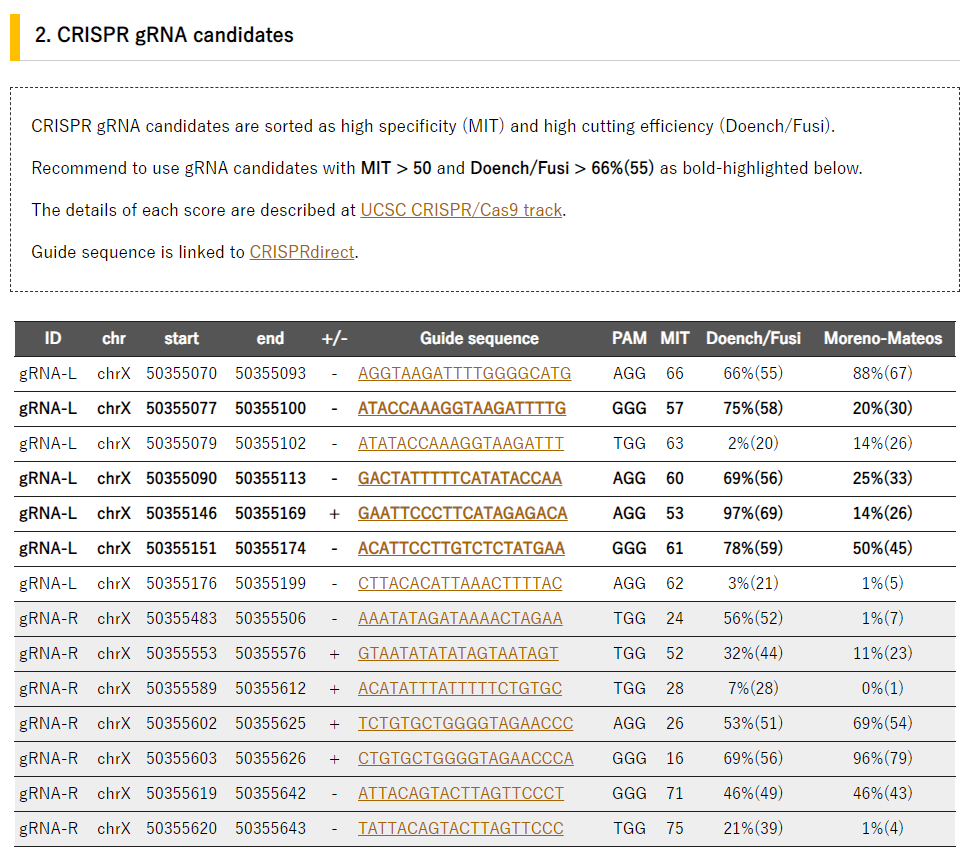
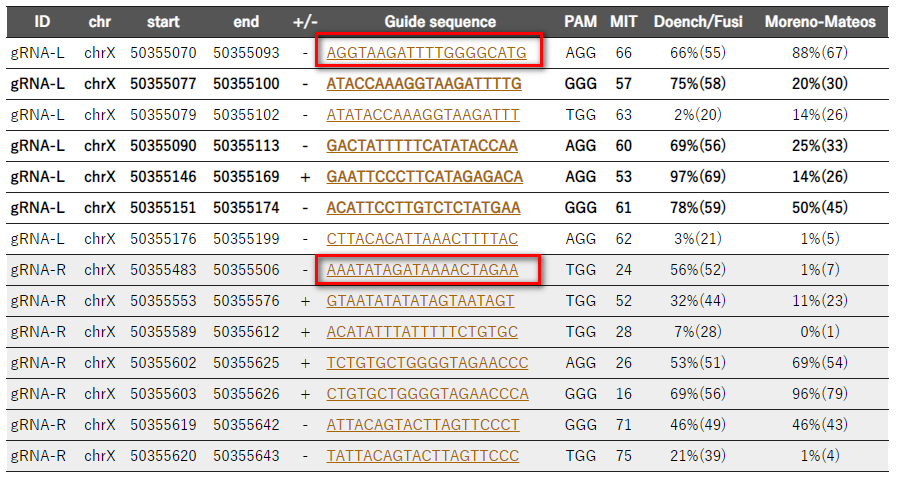
CRCISPR gRNA candidates には、下記の様にgRNA-LおよびgRNA-Rの具体的配列が表示されます。

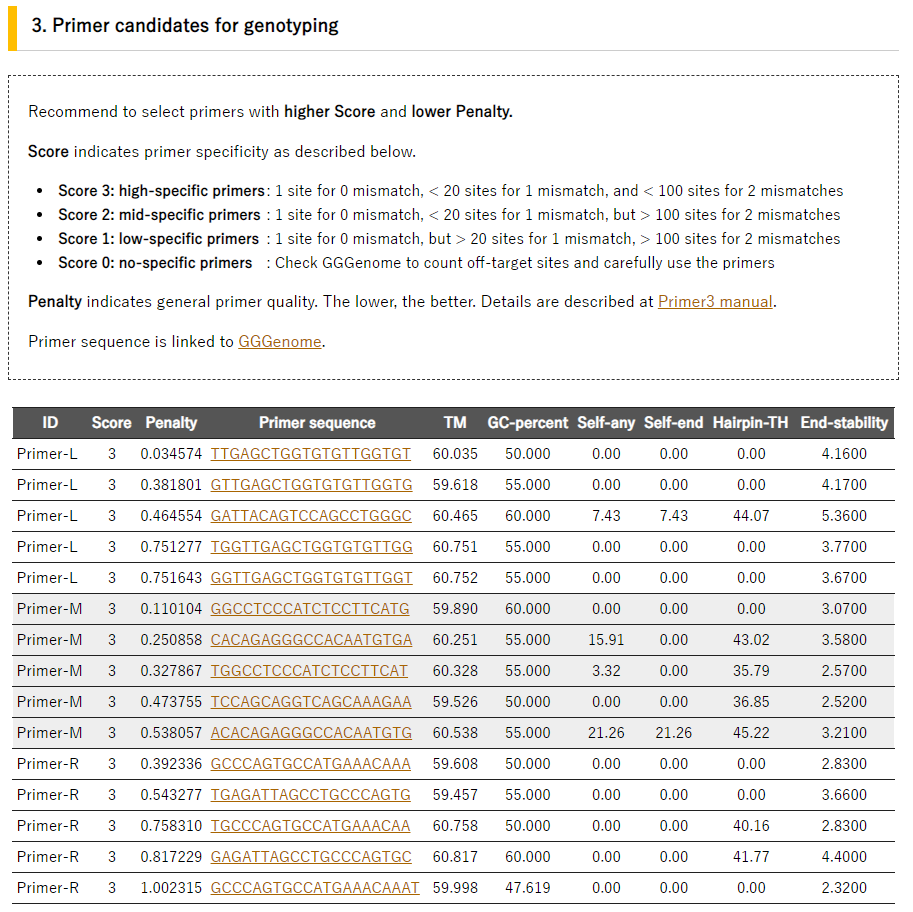
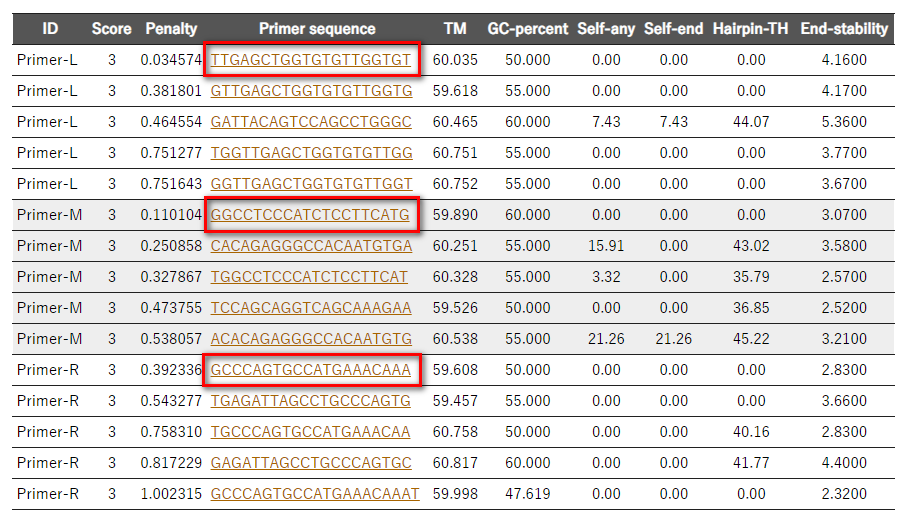
- 3. ジェノタイピングPCRプライマーの候補一覧
Primer candidates for genotyping には、下記の様にPrimer L、M、Rの具体的配列が表示されます。

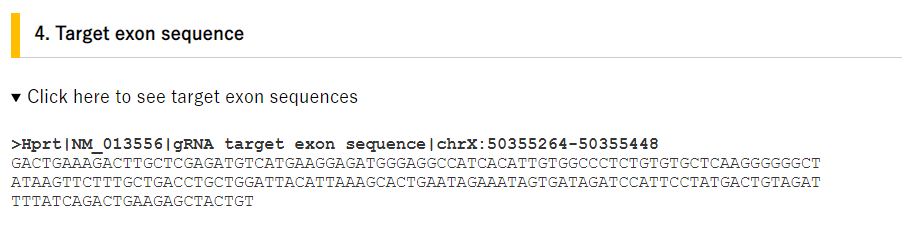
- 4. 切断されるエクソンの配列
Target exon sequence には、切断前のHPRTの配列が表示されます。
- 5. 切断後のmRNA配列
CDS after deletion には、切断前のHPRTの配列が表示されます。
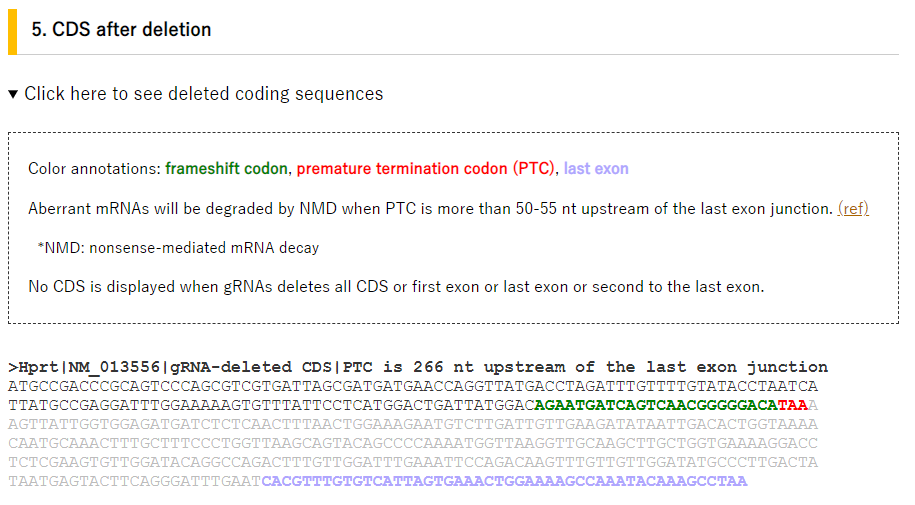
フレームシフトする配列や、フレームシフトによって発生する終止コドン、終止コドン後の配列等を確認できます。
「KOnezumi」の使用方法に関するお問い合わせは、筑波大学 久野 朗広先生までご連絡ください。
*下記リンクのInformation参照
http://www.md.tsukuba.ac.jp/LabAnimalResCNT/KOanimals/konezumi.html
KOnezumiから得られた配列をIDTで合成する
gRNAをIDTで合成する
取得したgRNAの情報をIDTで合成する方法を紹介します。
IDTのAlt-Rの特徴等についてはこちらのページをご参照ください。
■現在、初回30%オフキャンペーンも行っております。
IDTのAlt-Rの特徴等についてはこちらのページをご参照ください。
■現在、初回30%オフキャンペーンも行っております。
- IDTオーダーサイトにアクセスします。
URL:https://sg.idtdna.com/site - 右上のFlagを日本に変更し、サインインをクリックします。
- ログインもしくは新規登録をします。
※新規登録はこちらからお願いします。 - CRISPR-Cas9をクリックします。
このページのリンクはこちらです。 - [ORDER IN TUBES] ボタンからcrRNAを入力します。
- KOnezumiからgRNAの配列を選択します。
配列名と配列をコピー&ペーストで入力し、[Continue]をクリックします。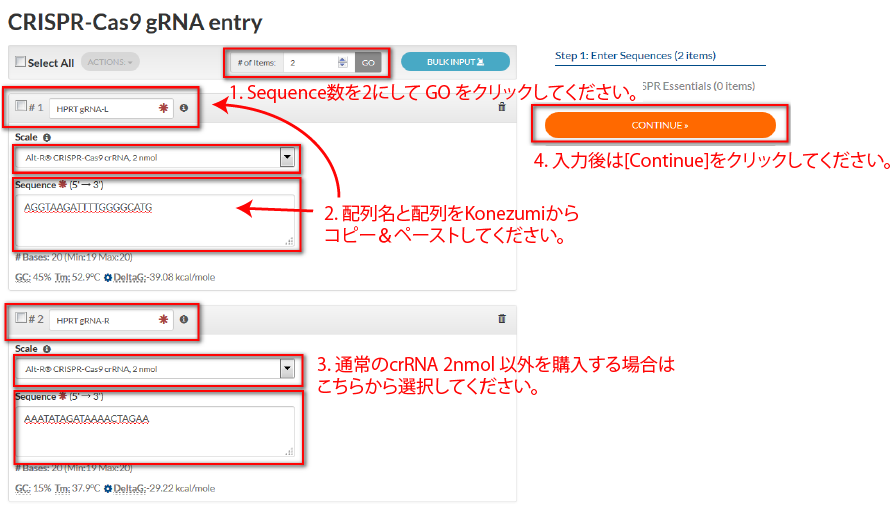
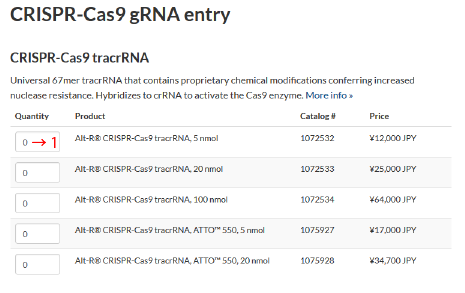
※配列名は、HPRT-1 gRNA-Lなど、遺伝子名&順位等がオススメです。 - tracrRNAと、必要であればCas9タンパク質もショッピングカートに入れます。
それぞれ必要なtracrRNA、Cas9の個数を入力します。

その後、[ADD TO ORDER] をクリックします。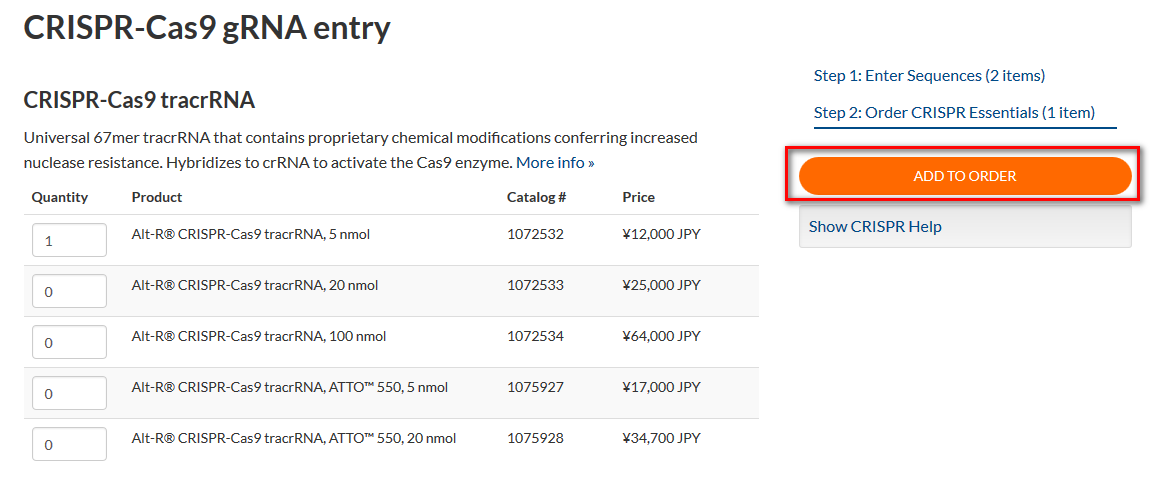
-
プライマーも購入される場合は、このまま「プライマーをIDTで合成する」にお進みください。
プライマーが必要ない場合、ショッピングカート以降はこちらをご参照ください。
プライマーをIDTで合成する
- IDTオーダーサイトにアクセスします。
URL:https://sg.idtdna.com/site - 右上のFlagを日本に変更し、サインインをクリックします。

- ログインもしくは新規登録をします。

※新規登録はこちらからお願いします。 - Custom DNA Oligosをクリックします。
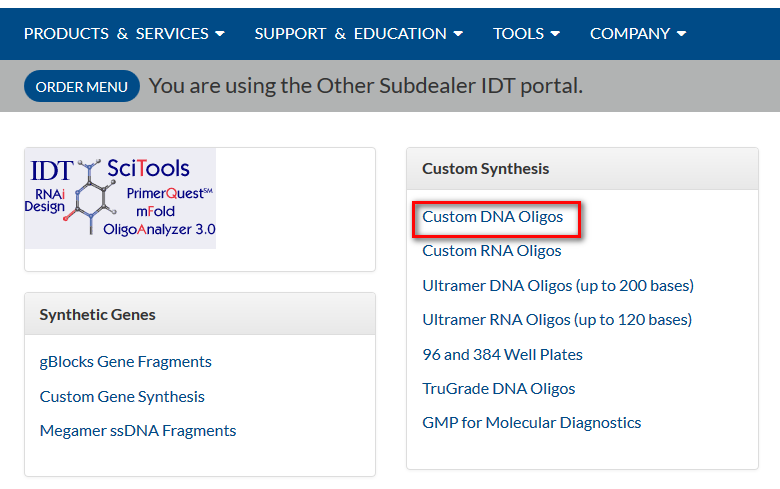
- KOnezumiからプライマーを選択します。
配列名を記入し、配列をコピー&ペーストします。
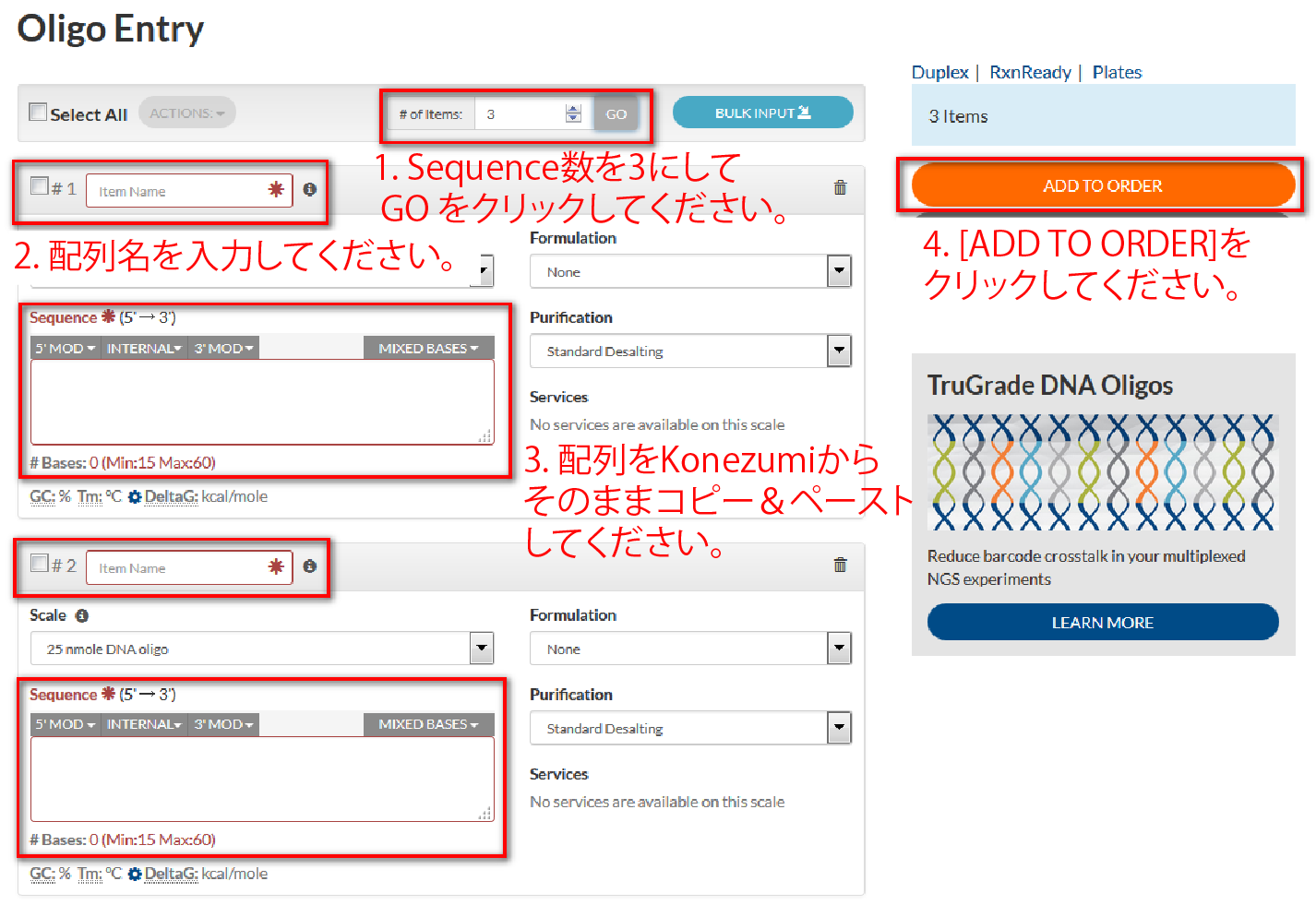
※配列名は、HPRT-1 Primer-Lなど、遺伝子名&順位等を加える事をオススメします。
製品フォーカス
Alt-R® CRISPR-Cas9 System
Alt-R® CRISPR-Cas9 Systemには、ゲノム編集を行うためのキーとなる試薬が揃っています。S. pyogenesの持つCRISPR-Cas9 systemに由来する本システムは、下記の様に多数のアドバンテージがあります。- 他社の手法と比較し、オンターゲット率が向上します。
- Cas9 RNPが正確にされます。
- リポフェクションやエレクトロポレーションで、効率よくRNPが導入できます。
- sgRNAやCas9 mRNAの様に、自然免疫を惹起しません。
Alt-R® CRISPR-Cpf1 System
Alt-R® CRISPR-Cpf1 Systemを用いると、これまでCRISPR-Cas9で切断できなかったサイトでゲノム編集を行えます。Cpf1でDNAを切断すると、5'突出末端となります。本試薬には下記の特徴があります。
- AT-richなゲノム領域でもゲノム編集が行えます。
- Cas9では切断できなかったサイトを補填できます。
- Cpf1 NucleaseとcrRNAのみで、ゲノム編集を行えます。tracrRNAは必要ありません。
- エレクトロポレーション法で効率よくRNPを導入できます。