簡便にゲノム編集の変異導入効率が測定できるT7EIアッセイとは
Alt-R® Genome Editing Detection Kit―PCRを用いるT7エンドヌクレアーゼI(T7EI)アッセイ
製品フォーカス:
PCRを用いるT7エンドヌクレアーゼI(T7EI)アッセイ、Alt-R® Genome Editing Detection Kitが、CRISPRで導入した変異の検出方法としてお勧めである理由をご説明します。
2017年3月30日
製品フォーカス:
PCRを用いるT7エンドヌクレアーゼI(T7EI)アッセイ、Alt-R® Genome Editing Detection Kitが、CRISPRで導入した変異の検出方法としてお勧めである理由をご説明します。
2017年3月30日
ゲノム編集の用途開発、試薬開発は急速に進んでいます。
CRISPR-Cas9、CRISPR-Cas12a(Cpf1)、その他のCRISPRシステムにおいて、実験結果の解釈にはオンターゲットに対するゲノム編集の確認が大切です。弊社は現在、ゲノム編集による変異の検出用T7EIアッセイには、Alt-R Genome Editing Detection Kitをお勧めしています(図1)。
この方法を用いると、きれいな電気泳動結果が得られるばかりでなく、一般的な分子生物学研究用の機器だけで実施でき、大半の場合、T7EIによる消化の前にPCR産物の精製をおこなう必要もありません。
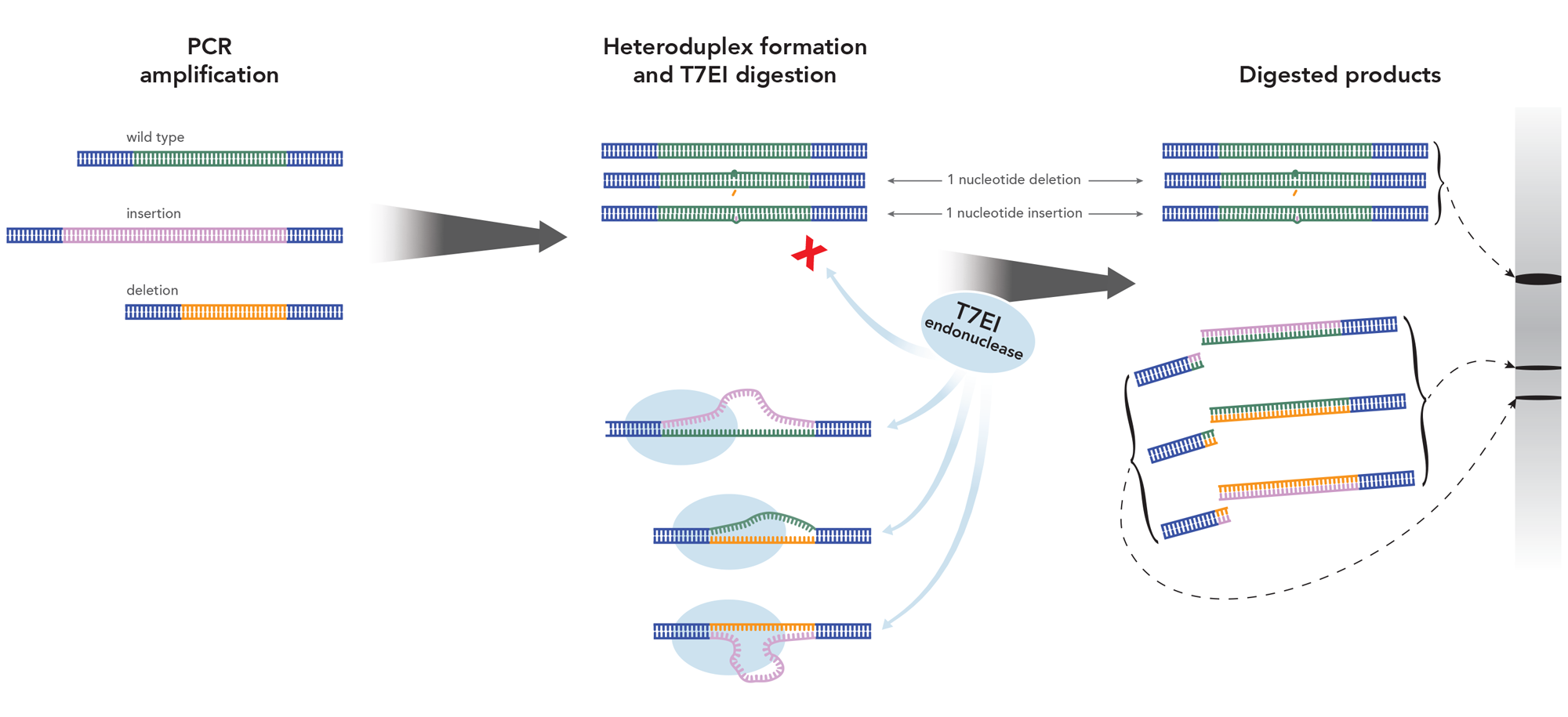
Alt-R Genome Editing Detection Kitには、T7EI酵素と緩衝液、T7EIポジティブコントロール反応用テンプレート-プライマーミックスが含まれていますが、ターゲットに対するPCRプライマーは設計する必要があります。 (PCRのデザインについては、 下記[Alt-R Genome Editing Detection Kit 使い方のヒント]をご覧ください。)
CRISPR-Cas9、CRISPR-Cas12a(Cpf1)、その他のCRISPRシステムにおいて、実験結果の解釈にはオンターゲットに対するゲノム編集の確認が大切です。弊社は現在、ゲノム編集による変異の検出用T7EIアッセイには、Alt-R Genome Editing Detection Kitをお勧めしています(図1)。
この方法を用いると、きれいな電気泳動結果が得られるばかりでなく、一般的な分子生物学研究用の機器だけで実施でき、大半の場合、T7EIによる消化の前にPCR産物の精製をおこなう必要もありません。

図1. Alt-R Genome Editing Detection Kitの基礎であるT7EIアッセイの概略図
PCRを用いて標的ゲノム領域を増幅します(注:PCR試薬はキットには含まれていません)。
サーマルサイクラーでPCR産物の変性・再アニールをおこない、野生型DNAと変異DNA間にヘテロ二本鎖が形成できるようにします。
再アニーリングしたPCR産物のうち、T7EIはミスマッチDNAヘテロ二本鎖のみを切断します。ヘテロ2本鎖を形成後、ゲルやキャピラリー電気泳動で結果を分析します。
Alt-R Genome Editing Detection Kitには、T7EI酵素と緩衝液、T7EIポジティブコントロール反応用テンプレート-プライマーミックスが含まれていますが、ターゲットに対するPCRプライマーは設計する必要があります。 (PCRのデザインについては、 下記[Alt-R Genome Editing Detection Kit 使い方のヒント]をご覧ください。)
2塩基以上の挿入・欠失に対するT7EIの特異性
T7EIは、ゲノム編集における非相同末端結合(NHEJ)活性によって生成される2塩基以上の挿入・欠失(indels)を認識します。T7EIは1塩基indelsを認識しないため、T7EIアッセイは全体の編集度を過小評価する可能性があります。
評価のばらつきは標的によって異なり、DNA切断後のNHEJ媒介修復の種類に影響されます(図2)。
蛍光活性化セルソーティング(FACS)を用いて、トランスフェクションされた細胞のみをエンリッチメントするために、弊社は蛍光標識tracrRNAも提供しています(tracrRNA-ATTO550)。
評価のばらつきは標的によって異なり、DNA切断後のNHEJ媒介修復の種類に影響されます(図2)。
蛍光活性化セルソーティング(FACS)を用いて、トランスフェクションされた細胞のみをエンリッチメントするために、弊社は蛍光標識tracrRNAも提供しています(tracrRNA-ATTO550)。

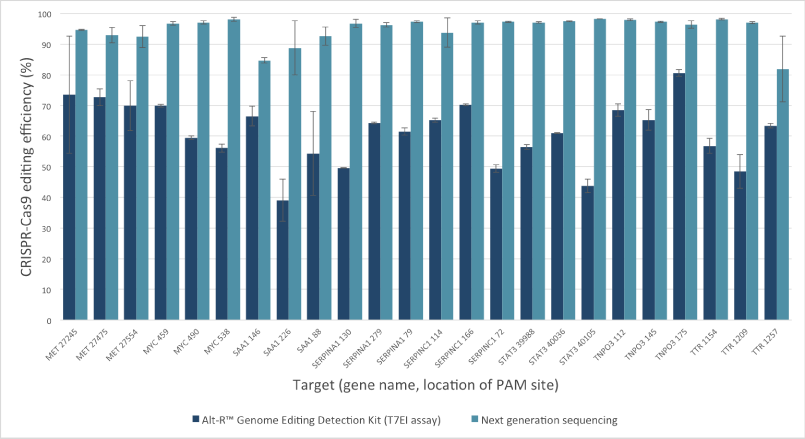
図2. T7EIミスマッチエンドヌクレアーゼアッセイによるゲノム編集効率の推定値と、次世代シーケンシングの編集効率
Streptococcus pyogenes Cas9を安定的に発現するHEK-293細胞に、リポフェクションによってAlt-R CRISPR-Cas9 RNAオリゴヌクレオチド(30nM)を導入しました。
8遺伝子のそれぞれにおいて、3つのサイトをターゲティングしました。Alt-R Genome Editing Detection Kit(Navy)を用いて、トランスフェクション細胞由来のゲノムDNAサンプルの試験を行い、編集効率を評価しました。
同じDNAサンプルを次世代シーケンシング(blue)でも分析しました。アンプリコンをMiSeqシステム(Illumina社)にかけ、オープンソースのデータ処理プログラム(Pinello L, Canver MC, et al. (2016) Nat Biotechnol 34:695– 697.)を用いて解析しました。
エラーバーは、3回おこなったリポフェクション実験の標準偏差を示しています。
なお、CRISPR-Cas9では次世代シーケンシングの編集効率の方がT7EIアッセイと比較して高い編集効率を示すのに対し、弊社がAlt-R CRISPR-Cas12a(Cpf1)実験で得た予備的な結果では、T7EIアッセイと次世代シーケンシングで同等の編集効率結果が得られることが示唆されています。
Cpf1エンドヌクレアーゼによる5'位の切断は突出末端で、また、おそらくはNHEJ修復により、Cas9エンドヌクレアーゼにより生じる平滑末端切断のNHEJ修復よりも、1塩基indelは少なくなるものと考えられます。
Alt-R Genome Editing Detection Kitの詳細とプロトコールについては、User guides and protocolsをご覧ください。
Cpf1エンドヌクレアーゼによる5'位の切断は突出末端で、また、おそらくはNHEJ修復により、Cas9エンドヌクレアーゼにより生じる平滑末端切断のNHEJ修復よりも、1塩基indelは少なくなるものと考えられます。
Alt-R Genome Editing Detection Kitの詳細とプロトコールについては、User guides and protocolsをご覧ください。
Alt-R Genome Editing Detection Kit 使い方のコツ
■ PCRデザイン
CRISPR切断部位の両側に100 bp以上の余剰配列がある、長さ600 bp~1000 bpのPCRアンプリコンを使用することをお勧めします。CRISPR切断部位を中心から外し、ゲル上で明確な消化産物サイズが2つ出るようにします。
PCR条件を最適化し、ゲノム編集をおこなう前に、ゲノムDNAから単一のPCR産物が増幅されることを電気泳動で確認します。
PrimerQuest®ツールを用いると、簡単にPCRアッセイをデザインできます。
ご利用方法の紹介(日本語)はこちら
■ Alt-R CRISPR-Cas9 System
Alt-R CRISPR-Cas9 tracrRNA-ATTO™ 550は、Cas9ヌクレアーゼを用いたゲノム編集実験で、非標識のtracrRNAと置き換えることができる蛍光標識tracrRNAです。
この蛍光色素によって、蛍光活性化セルソーティング(FACS)分析を用いてトランスフェクションされた細胞をエンリッチすることができるようになります。
ゲノム編集が行われた細胞を、Alt-R Genome Editing Detection Kitを使って同定できる確率が高くなります。
FACsでソーティングした際のゲノム編集効率については、こちらの図6に掲載しております。
CRISPR切断部位の両側に100 bp以上の余剰配列がある、長さ600 bp~1000 bpのPCRアンプリコンを使用することをお勧めします。CRISPR切断部位を中心から外し、ゲル上で明確な消化産物サイズが2つ出るようにします。
PCR条件を最適化し、ゲノム編集をおこなう前に、ゲノムDNAから単一のPCR産物が増幅されることを電気泳動で確認します。
PrimerQuest®ツールを用いると、簡単にPCRアッセイをデザインできます。
ご利用方法の紹介(日本語)はこちら
■ Alt-R CRISPR-Cas9 System
Alt-R CRISPR-Cas9 tracrRNA-ATTO™ 550は、Cas9ヌクレアーゼを用いたゲノム編集実験で、非標識のtracrRNAと置き換えることができる蛍光標識tracrRNAです。
この蛍光色素によって、蛍光活性化セルソーティング(FACS)分析を用いてトランスフェクションされた細胞をエンリッチすることができるようになります。
ゲノム編集が行われた細胞を、Alt-R Genome Editing Detection Kitを使って同定できる確率が高くなります。
FACsでソーティングした際のゲノム編集効率については、こちらの図6に掲載しております。
References
原文:A simple method to detect on-target editing or measure genome editing efficiency in CRISPR experiments
著者:Maureen Young, PhD, Senior Scientific Writer, IDT. 翻訳:安井 孝彰
著者:Maureen Young, PhD, Senior Scientific Writer, IDT. 翻訳:安井 孝彰
製品フォーカス
Alt-R® CRISPR-Cas9 System
Alt-R® CRISPR-Cas9 Systemには、ゲノム編集を行うためのキーとなる試薬が揃っています。S. pyogenesの持つCRISPR-Cas9 systemに由来する本システムは、下記の様に多数のアドバンテージがあります。- 他社の手法と比較し、オンターゲット率が向上します。
- Cas9 RNPが正確にされます。
- リポフェクションやエレクトロポレーションで、効率よくRNPが導入できます。
- sgRNAやCas9 mRNAの様に、自然免疫を惹起しません。
Alt-R® CRISPR-Cpf1 System
Alt-R® CRISPR-Cpf1 Systemを用いると、これまでCRISPR-Cas9で切断できなかったサイトでゲノム編集を行えます。Cpf1でDNAを切断すると、5'突出末端となります。本試薬には下記の特徴があります。
- AT-richなゲノム領域でもゲノム編集が行えます。
- Cas9では切断できなかったサイトを補填できます。
- Cpf1 NucleaseとcrRNAのみで、ゲノム編集を行えます。tracrRNAは必要ありません。
- エレクトロポレーション法で効率よくRNPを導入できます。