xGen® Lockdown® Probesを用いたFFPEサンプルのDNAエンリッチメント
確実なシーケンスデータ取得のための推奨事項
ハイライト
• xGen® Lockdown® Probesを用いた、ホルマリン固定パラフィン包埋(FFPE)組織ブロックからのDNA抽出、DNAの質の評価、ターゲットエンリッチメント用ライブラリーの調製に関する推奨事項
• アーカイブ組織サンプルのカバレッジ・体細胞変異コール感度向上のガイドライン
• xGen® Lockdown® Probesを用いた、ホルマリン固定パラフィン包埋(FFPE)組織ブロックからのDNA抽出、DNAの質の評価、ターゲットエンリッチメント用ライブラリーの調製に関する推奨事項
• アーカイブ組織サンプルのカバレッジ・体細胞変異コール感度向上のガイドライン
Introduction
癌ゲノムは通常、FFPEサンプルを用いて研究が行われています。FFPEサンプル中には多様のクローン集団が存在し、各クローンで特有のドライバー変異をもつため、すべてのクローンで解析が行われないと正確な評価が出来ません。
さらに、このようなサンプルの腫瘍DNA含有率は低く、特性を解析するには感度の高い変異検出能力が必要です。
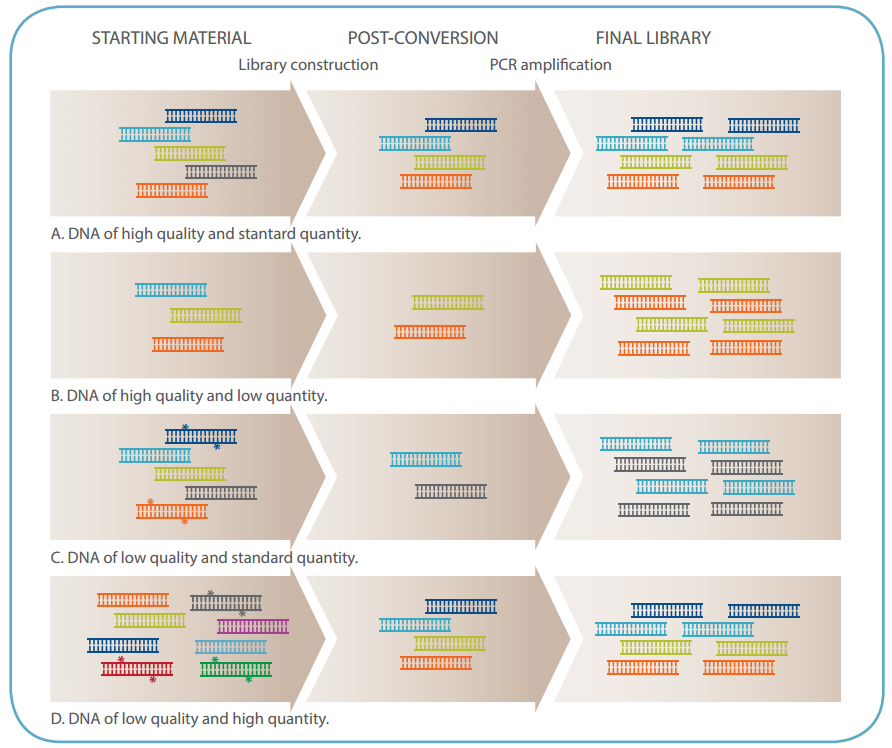
そのため、腫瘍の特徴を正確に評価するには、ライブラリーが十分多様であること、サンプルをバランスよく含んでいること、ユニークなシーケンシングのカバレッジが高いことが要求されます(図1)。
xGen Lockdown Probesを用いてサンプルのエンリッチメントを行うと、最低限のシーケンシングリードで、ターゲット領域のカバレッジを深くすることができます。
ただし、FFPE DNAの質と量は多様であるため、サンプルの種類を適切に評価、処理してライブラリーの複雑性を最大化し、ターゲットのカバレッジを高めることが大切です。
このアプリケーションノートでは、市販のDNA品質評価キットを用いてFFPE DNAから最終ライブラリーの複雑性を推定し最大化する方法について説明します。
さらに、このようなサンプルの腫瘍DNA含有率は低く、特性を解析するには感度の高い変異検出能力が必要です。
そのため、腫瘍の特徴を正確に評価するには、ライブラリーが十分多様であること、サンプルをバランスよく含んでいること、ユニークなシーケンシングのカバレッジが高いことが要求されます(図1)。
xGen Lockdown Probesを用いてサンプルのエンリッチメントを行うと、最低限のシーケンシングリードで、ターゲット領域のカバレッジを深くすることができます。
ただし、FFPE DNAの質と量は多様であるため、サンプルの種類を適切に評価、処理してライブラリーの複雑性を最大化し、ターゲットのカバレッジを高めることが大切です。
このアプリケーションノートでは、市販のDNA品質評価キットを用いてFFPE DNAから最終ライブラリーの複雑性を推定し最大化する方法について説明します。
Results
さまざまな腫瘍組織FFPEブロック(Asterand BioSciences社)から、DNAを抽出しました。
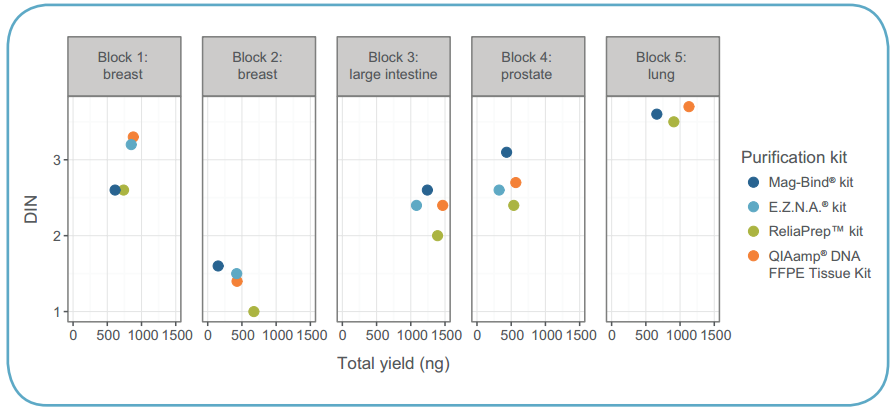
市販の4種類の抽出キットで行ったDNAの質・収量の測定結果は一貫していました(図2)。
このデータは、DNAの質と収量がDNA抽出キットとは無関係であることを示しており、質と収量はFFPEブロックの最初の固定や状態の影響を受けることがわかります。
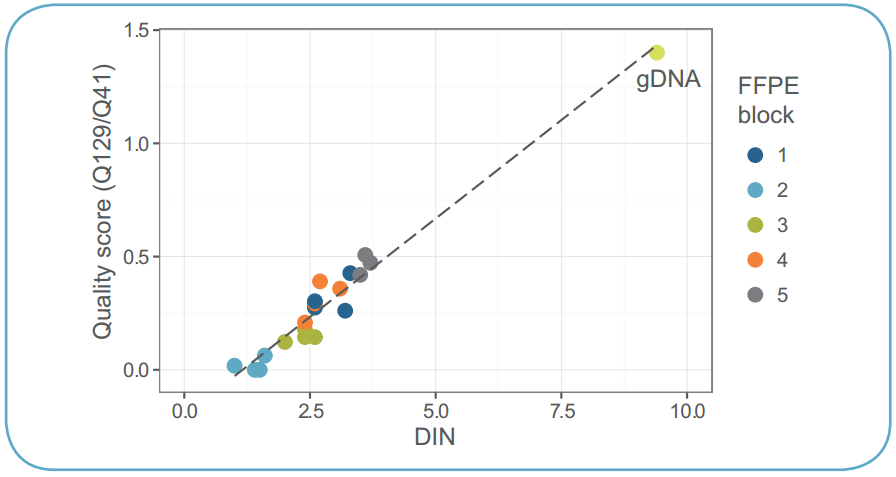
次に、DNAの質を評価する各種の方法について、どのようなサンプルの状態から、有用で信頼できるシーケンスデータが得られるか検討しました(図3、表1)。
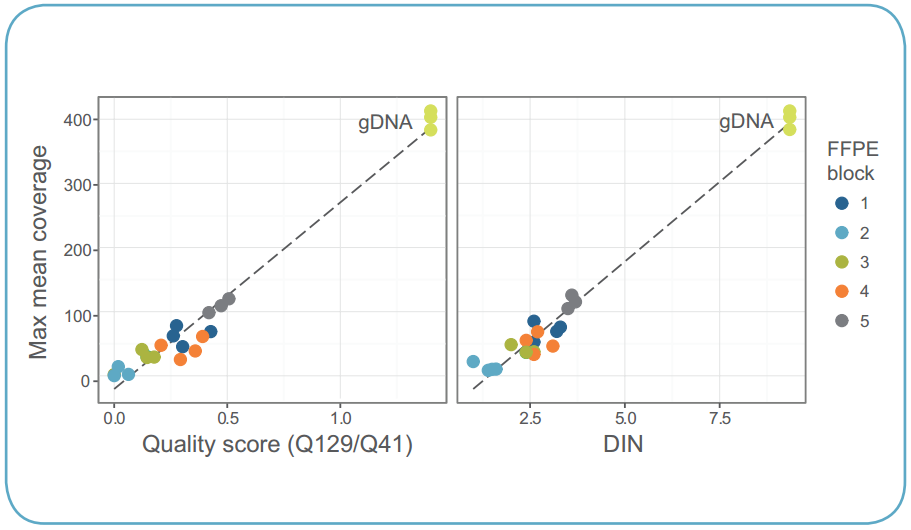
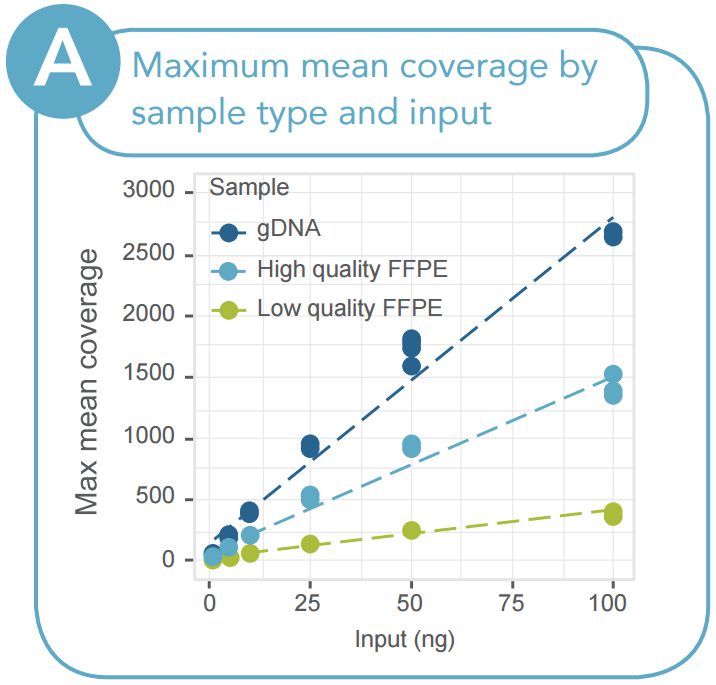
図4は、平均カバレッジの最大値を、サンプル品質のスコアまたはDNA分解度(DNA Integrity Number、DIN)を用いて測定した場合の、比較結果を示しています。
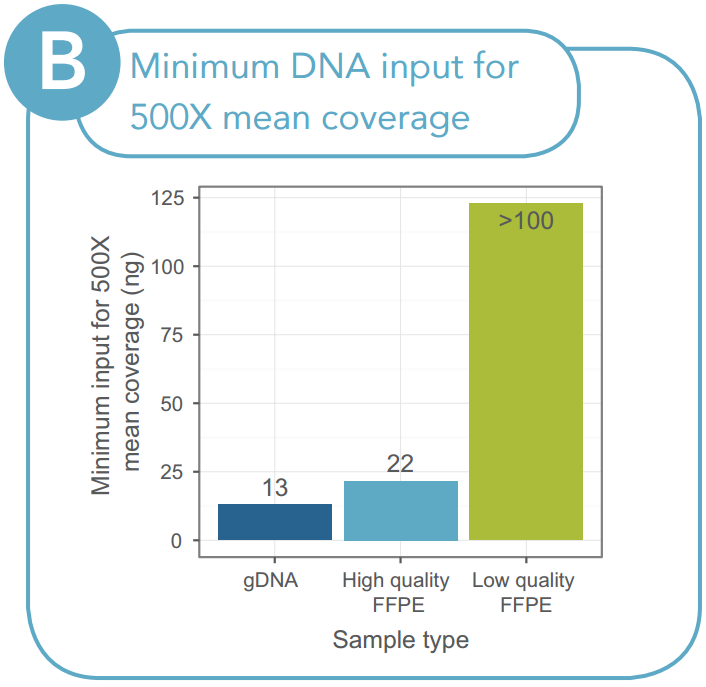
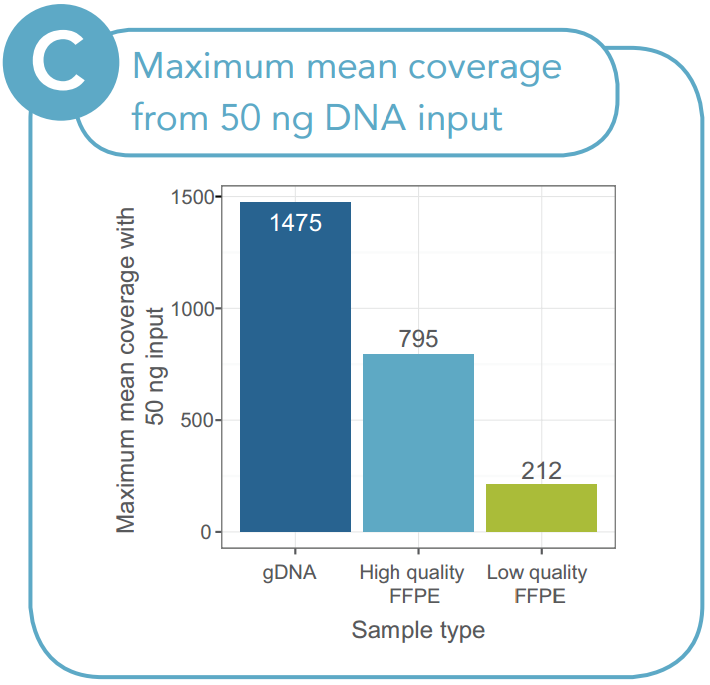
最後に、コントロールgDNAと質の高いFFPEおよび質の低いFFPEのインプット量を滴定し(図5)、インプット量と最終産物の複雑性の関係を明らかにしました。これらのデータは、最初のサンプル量を増やすことによって、一部のFFPEライブラリーの「レスキュー」ができることを示しています。
■ DNAのインプット量がライブラリーの複雑性に及ぼす影響
表1.FFPE組織から単離されたDNAの品質の評価
品質のスコア(Qスコア)を、129bp/41bpアンプリコン比として表しました。DNAの分解が進むとテンプレートの長いサンプルでPCR増幅できないため、Qスコアが低くなり、サンプルの品質が悪いと判断できます。理想的なQスコアは1です。これは、両アンプリコンの増幅が同等であり、DNAが分解されていないことを示しています。
表2. サンプルの種類およびインプット量ごとの、プレキャプチャー時PCR増幅サイクル
市販の4種類の抽出キットで行ったDNAの質・収量の測定結果は一貫していました(図2)。
このデータは、DNAの質と収量がDNA抽出キットとは無関係であることを示しており、質と収量はFFPEブロックの最初の固定や状態の影響を受けることがわかります。
次に、DNAの質を評価する各種の方法について、どのようなサンプルの状態から、有用で信頼できるシーケンスデータが得られるか検討しました(図3、表1)。
図4は、平均カバレッジの最大値を、サンプル品質のスコアまたはDNA分解度(DNA Integrity Number、DIN)を用いて測定した場合の、比較結果を示しています。
最後に、コントロールgDNAと質の高いFFPEおよび質の低いFFPEのインプット量を滴定し(図5)、インプット量と最終産物の複雑性の関係を明らかにしました。これらのデータは、最初のサンプル量を増やすことによって、一部のFFPEライブラリーの「レスキュー」ができることを示しています。
■ DNAのインプット量がライブラリーの複雑性に及ぼす影響
| サンプル名 | Q129/41 | DIN |
|---|---|---|
| 質の低いFFPE | < 0.2 | < 2.5 |
| 質の高いFFPE | < 0.4 | > 3.5 |
表1.FFPE組織から単離されたDNAの品質の評価
品質のスコア(Qスコア)を、129bp/41bpアンプリコン比として表しました。DNAの分解が進むとテンプレートの長いサンプルでPCR増幅できないため、Qスコアが低くなり、サンプルの品質が悪いと判断できます。理想的なQスコアは1です。これは、両アンプリコンの増幅が同等であり、DNAが分解されていないことを示しています。
| サンプルの種類 | 入力量 (ng) | |||||
|---|---|---|---|---|---|---|
| 1 | 5 | 10 | 25 | 50 | 100 | |
| gDNA | 14 | 12 | 11 | 8 | 7 | 6 |
| FFPE | 16 | 14 | 12 | 9 | 8 | 7 |
表2. サンプルの種類およびインプット量ごとの、プレキャプチャー時PCR増幅サイクル
結論および最終的な推奨事項
今回、FFPEサンプルから抽出DNAの品質と量がシーケンシングライブラリーの複雑性にどのように影響するかを検討しました。
検討した理由は、抽出DNAの品質と量は保存されたサンプルの取り扱い時に最も懸念される要因であるためです。今回のデータで次のことがわかりました。
GC含量の多い領域が取得しにくいのはプローブの合成が難しいためですが、IDTでは、プローブ1本ずつ全てにおいて品質管理を行っているため、納品したプローブプール内に各プローブが確実に必要量含まれています。そのため、GCリッチな領域を含むターゲット領域全体を均一に取得可能です。
» xGen®製品の特徴について詳しくはこちらをご覧下さい。
診断用のxGen Lockdownプローブも利用できます。 詳細は、japan-cc@idtdna.comにお問い合わせください。
» xGen® Lockdown® Probesの詳細についてはこちらをご覧ください。
» xGen® Lockdown® Panelsの詳細については、こちらをご覧ください。
» xGen® Universal Blockers – TS Mixの詳細については、こちらをご覧ください。
また、サンプル量により、得られるデータの品質がどのように変わるかも併せて紹介しています。
検討した理由は、抽出DNAの品質と量は保存されたサンプルの取り扱い時に最も懸念される要因であるためです。今回のデータで次のことがわかりました。
- FFPEサンプルから抽出されたDNAの品質と収量は、抽出キットとは無関係です。むしろ、保存中・固定中の取り扱い要因が関係している可能性が大きいです。
- ライブラリー調製前のDNAの品質の評価では、品質のスコア(Qスコア、129bp/41bpアンプリコン比)とDNA分解度(DIN)は相関するため、QスコアやDINを用いて最終ライブラリーの複雑性を予測することができます。
今回、KAPA hgDNA Quantification and QC KitによるQスコアの測定値が0.4を超えるDNA、あるいはAgilent 4200 TapeStationシステムによるDNA分解度(DIN)の測定値が3.5を超えているDNAは、信頼性の高いシーケンスデータを取得するのに必要な、複雑性の高いライブラリーの調製に十分な品質をもっているという知見を得ました。 - FFPEサンプルから抽出したDNAを用いて、十分な品質のエンリッチメントしたライブラリーを調製することができます。
しかしながら、組織サンプルの取り扱い中・保存中に変動が生じるため、目的のカバレッジに必要な最小限のインプット量を求めるには、FFPE DNAの品質の管理(QC)を行うことが重要です。
シーケンシング結果はDNAの品質とインプット量によって異なってきます。
References
原文: DNA enrichment from formalin-fixed, paraffin-embedded
(FFPE) samples using xGen® Lockdown® Probes
翻訳・監修:浜本 雄次
翻訳・監修:浜本 雄次
製品フォーカス
xGen® 製品の特徴
xGen® 製品は、高品質なプローブを1本ずつ合成し、全てのプローブに品質管理及び納品量の標準化を行っています。SNPs、インデル、CNV、LOH、および転移の高感度の検出に有用で、既存のパネルを拡張したり、完全にカスタマイズされたパネルを作成する柔軟性も持ち合わせています。GC含量の多い領域が取得しにくいのはプローブの合成が難しいためですが、IDTでは、プローブ1本ずつ全てにおいて品質管理を行っているため、納品したプローブプール内に各プローブが確実に必要量含まれています。そのため、GCリッチな領域を含むターゲット領域全体を均一に取得可能です。
» xGen®製品の特徴について詳しくはこちらをご覧下さい。
診断用のxGen Lockdownプローブも利用できます。 詳細は、japan-cc@idtdna.comにお問い合わせください。
xGen® Lockdown® Probes
新規プローブセットの作製や、お手持ちのプローブセットの拡張・補填など実験目的に合わせたプローブセットをご購入いただけます。各遺伝子から目的の遺伝子をお選びいただくプレデザイン品と、ご希望のプローブ配列を合成するカスタム品をご用意しております。» xGen® Lockdown® Probesの詳細についてはこちらをご覧ください。
xGen® Lockdown® Panels
- エクソームおよび各疾患用に最適化されたプローブセットです。
- ・ xGen® Exome Research Panel - エクソームシーケンス用プローブセット
- ・ xGen® Acute Myeloid Leukemia Cancer Panel - AML(急性骨髄性白血病)用プローブセット
- ・ xGen® Pan-Cancer Panel - 12種のがんに共通して変異が見られる127遺伝子のプローブセット
- ・ xGen® Inherited Diseases Panel - HGMD®(Human Gene Mutation Database)に基づいた遺伝性疾患に関わる領域及びSNPsのプローブセット
- ・ xGen® Human ID Research Panel - ヒトDNAの識別用に設計されたプローブセット
- ・ xGen® Human mtDNA Research Panel - ヒトミトコンドリアDNAを網羅的に解析可能なプローブセット
- ・ xGen® CNV Backbone Panel—Tech Access - ヒトゲノム全体のCNV(コピー数多型)を解析するプローブセット
» xGen® Lockdown® Panelsの詳細については、こちらをご覧ください。
xGen® Universal Blocking Oligos
アダプター配列同士の結合を防ぐブロッキングオリゴです。xGen® Universal Blockers – TS MixはIllumina社のインデックスアダプター用に設計されており、シングルインデックスとデュアルインデックス用のどちらのタイプにもready-to-useで使用可能です。3’末端のC3スペーサー付加など非特異な配列の結合・増幅を防ぐ修飾がされており、オンターゲット率を大幅に向上させます。» xGen® Universal Blockers – TS Mixの詳細については、こちらをご覧ください。
Additional reading
xGen® Dual Index UMI Adaptersによるインデックスホッピングの解消と低頻度変異の検出
インデックスホッピングや低頻度変異の検出に有用なDual Index Apadtersについての説明と、このアダプターを用いて得られた具体的なで実験データを開示しています。プローブキャプチャーによるマルチプレックスに必要なサンプル量とデータの品質
プローブキャプチャー法でマルチプレックスを行った際に、一度にマルチプレックスするサンプル数によるデータの品質がどのように影響するか、を実際の実験データを用いて紹介しています。また、サンプル量により、得られるデータの品質がどのように変わるかも併せて紹介しています。