xGen® Dual Index UMI Adaptersによるインデックスホッピングの解消と低頻度変異の検出
Introduction
分子バーコード(Unique Molecular Identifier: UMI)を用いて設計されたNGSアダプター配列をユニークなデュアルインデックス配列と組み合わせると、扱いが難しいサンプル(低品質・FFPE等)や、少量しか使えないサンプルでも低頻度変異をより正確に検出することが可能です。
UMIはアダプター配列内のランダム配列であり、この配列によってサンプル中の各分子にそれぞれユニークなタグが付加されます。また、ユニークデュアルインデックスは、個々のサンプルに対していずれもユニークな2種類のインデックス配列 (P5およびP7) を組み合わせたものです。
これらのアダプター配列は市販のライゲーションベースのライブラリー構築キットに対して適合性があります。シングルインデックス(1)、デュアルインデックス(2)、UMI(3)を用いたデュアルインデックスのいずれかとして分析できます(図1)。
UMIはアダプター配列内のランダム配列であり、この配列によってサンプル中の各分子にそれぞれユニークなタグが付加されます。また、ユニークデュアルインデックスは、個々のサンプルに対していずれもユニークな2種類のインデックス配列 (P5およびP7) を組み合わせたものです。
これらのアダプター配列は市販のライゲーションベースのライブラリー構築キットに対して適合性があります。シングルインデックス(1)、デュアルインデックス(2)、UMI(3)を用いたデュアルインデックスのいずれかとして分析できます(図1)。
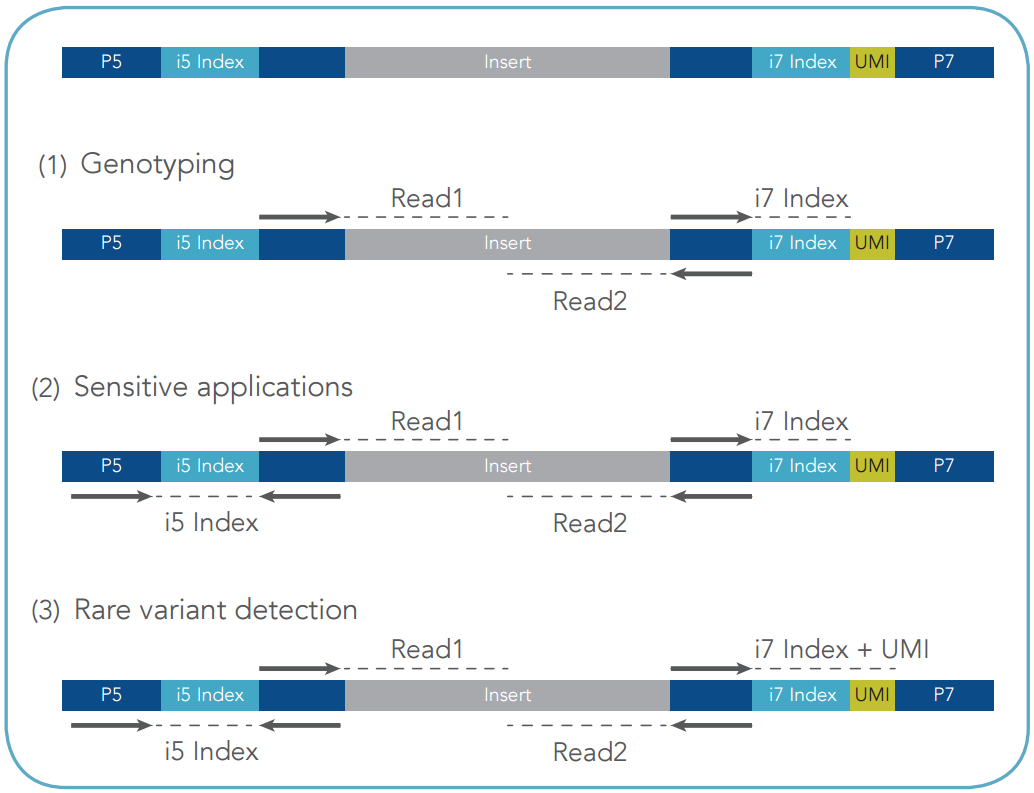 図1. UMIをもつユニークなデュアルインデックスアダプター配列は、アッセイの必要感度に応じて、3通りの方法のいずれかで読み取ることができます。 |
Technical Review
変異検出試験の方法
特性が十分に解明されているGenome in a Bottle(GIAB)細胞株や、個々の遺伝子型が決定されている腫瘍由来FFPEサンプルを用いてサンプル混合物を調製し、1%のマイナーアレル頻度状態を模擬的に作り出しました。
ライブラリー作製後、288個の共通SNPをターゲットとする長さ75 KBのカスタムxGen Lockdown Panelを用いて、全サンプルについてシングルプレックスによるキャプチャーを行いました。
変異コールはGIABにおいて信頼性の高い領域を、VarDictによるstart/stopの重複除去やUMIのコンセンサス解析により行いました。
表1.細胞株およびFFPE DNAによるアダプター配列の性能評価に用いたサンプル混合物の詳細
ライブラリー作製後、288個の共通SNPをターゲットとする長さ75 KBのカスタムxGen Lockdown Panelを用いて、全サンプルについてシングルプレックスによるキャプチャーを行いました。
変異コールはGIABにおいて信頼性の高い領域を、VarDictによるstart/stopの重複除去やUMIのコンセンサス解析により行いました。
表1.細胞株およびFFPE DNAによるアダプター配列の性能評価に用いたサンプル混合物の詳細
| サンプル情報 | 細胞株 | FFPE |
|---|---|---|
| サンプルソース | 99% NA12878/1% NA24835 | 99%乳癌/1%胃癌 |
| 代替SNP数 | 10 (1%)/44 (0.5%) | 20 (1%)/56 (0.5%) |
| ライブラリー構築時の入力量 | 25 ng | 25/50/100 ng |
| リード総数 | 約1800万 | 1200/2000/3800万 |
UMIを用いた変異検出の結果
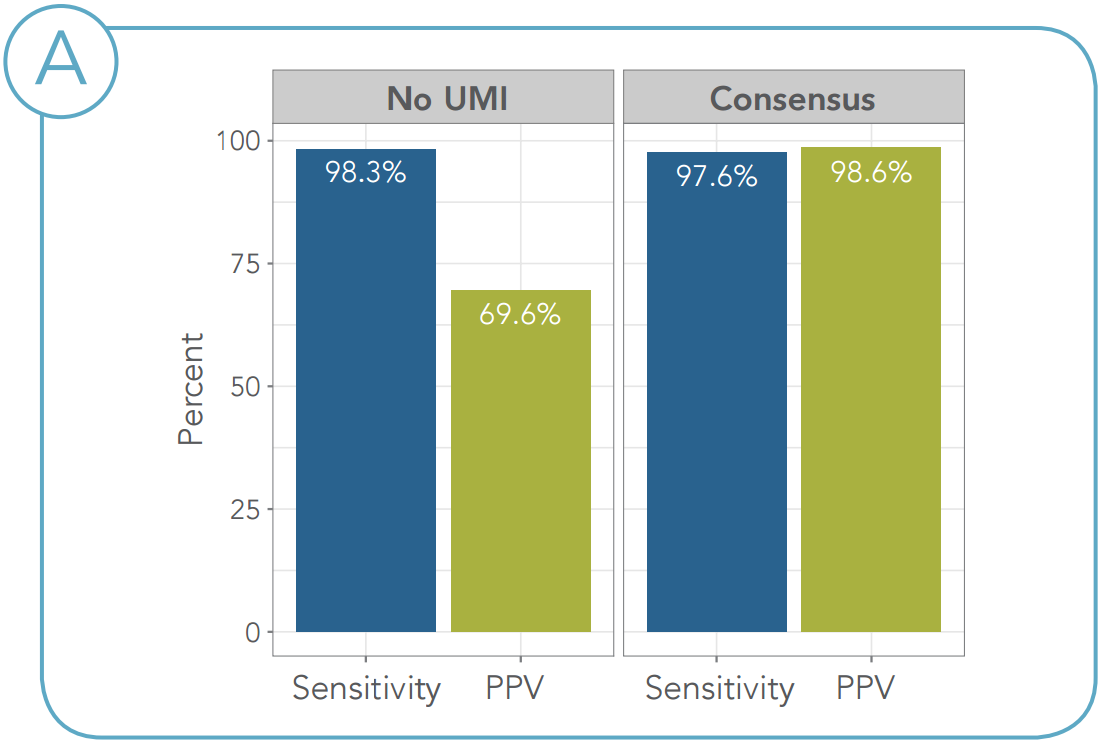 |
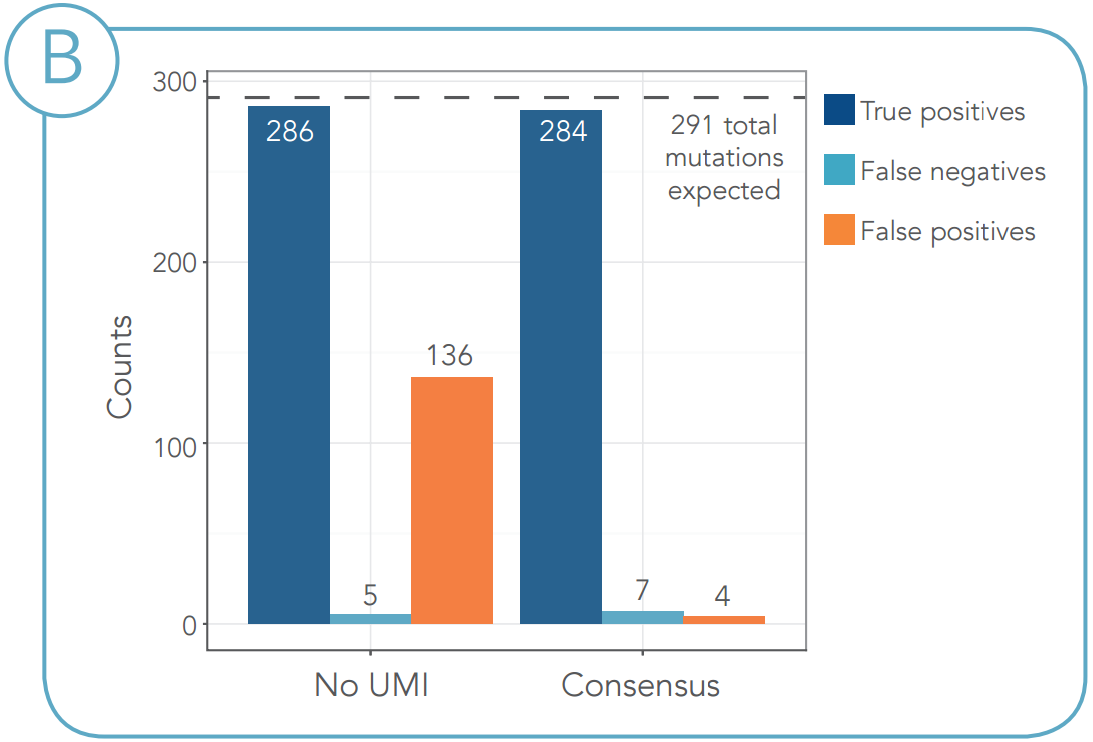 図4. 細胞株サンプルにおけるUMIによるエラーの修正 (A)UMIコンセンサス配列コールでPPVが69.6%から98.6%に向上しました。感度に対する影響は非常に小さかった。 (B)UMIコンセンサス配列コールを用いたところ、偽陽性コールの総数は136から4に減少しました。(PPV =予測値陽性) |
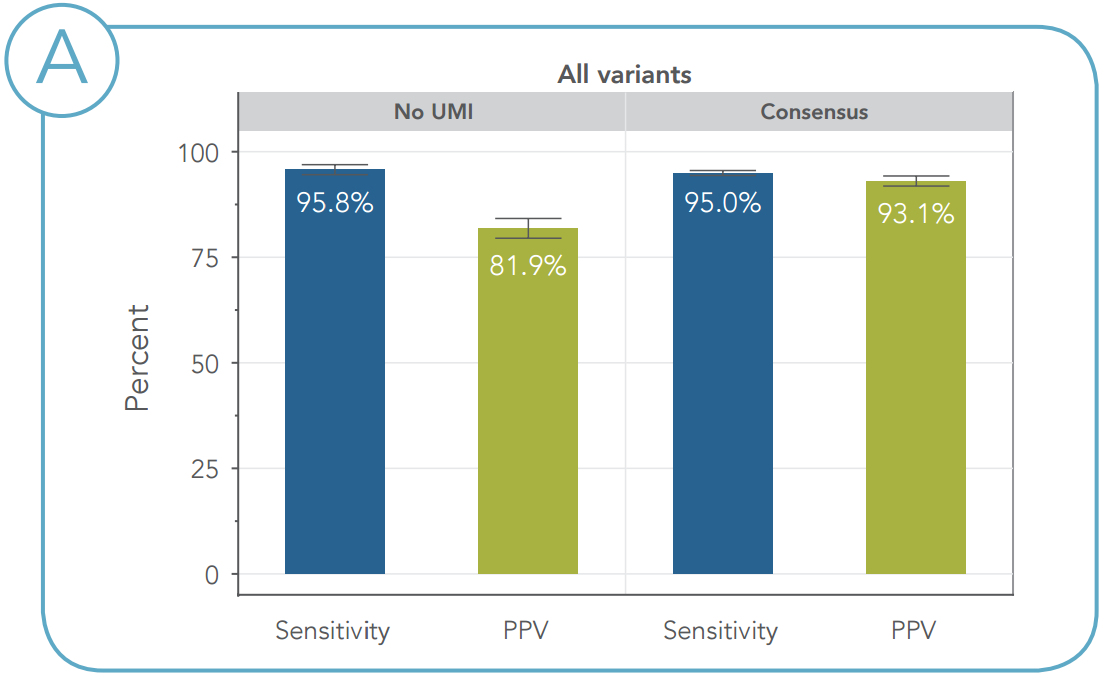 |
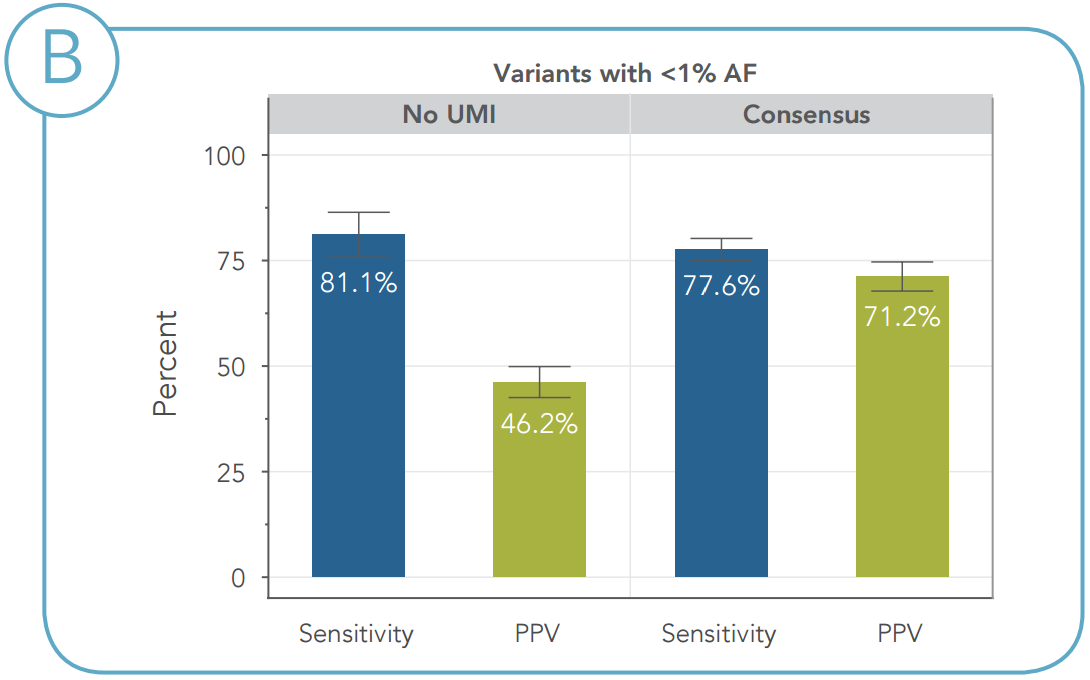 図5. FFPEサンプルにおけるUMIによるエラーの修正 (A)グラフは、全変異(n=340)について、最小アレル頻度0.6%を用いた場合の感度とPPVを示しています。 (B)グラフは、1%未満存在する変異(n=76)について、最小アレル頻度0.6%を用いた場合の感度とPPVを示しています。(AF=アレル頻度) |
FFPEサンプルDNAのカバレッジ結果
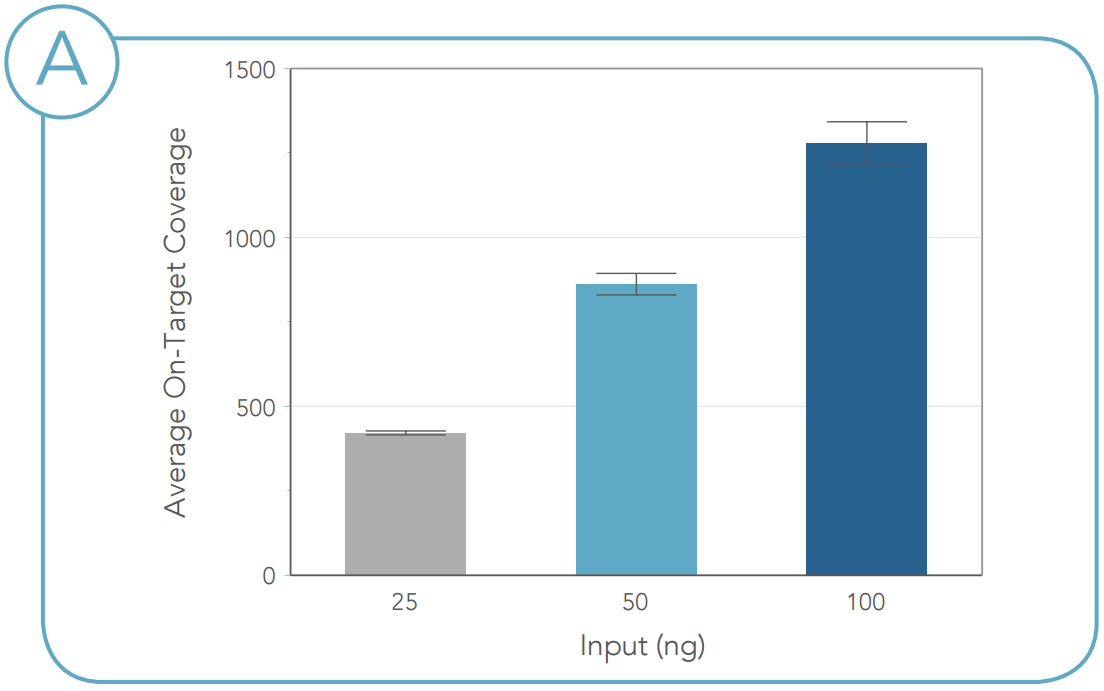 |
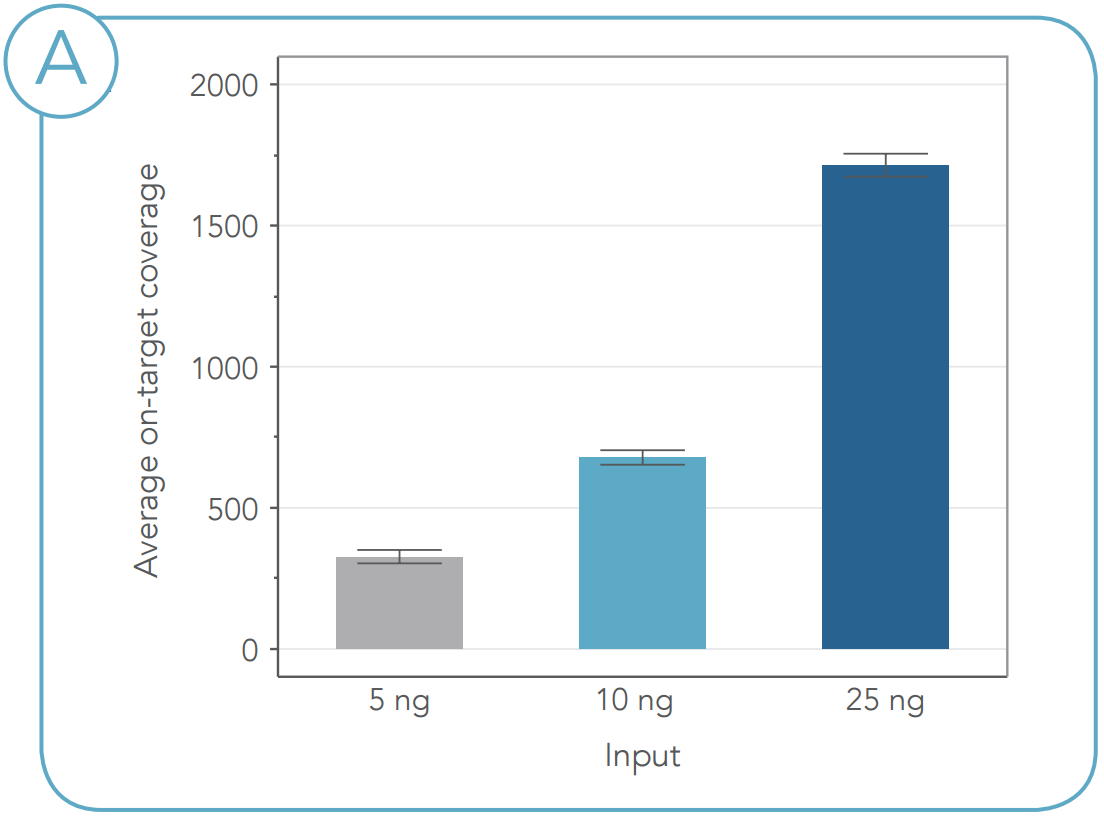
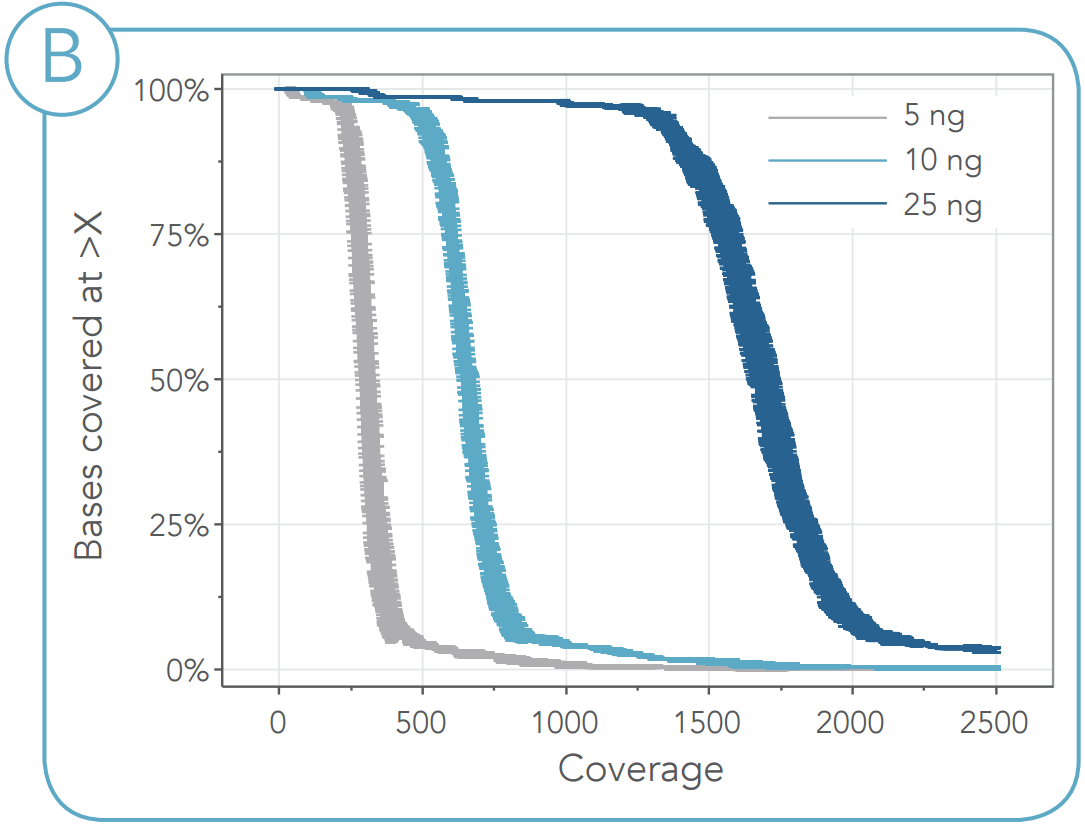
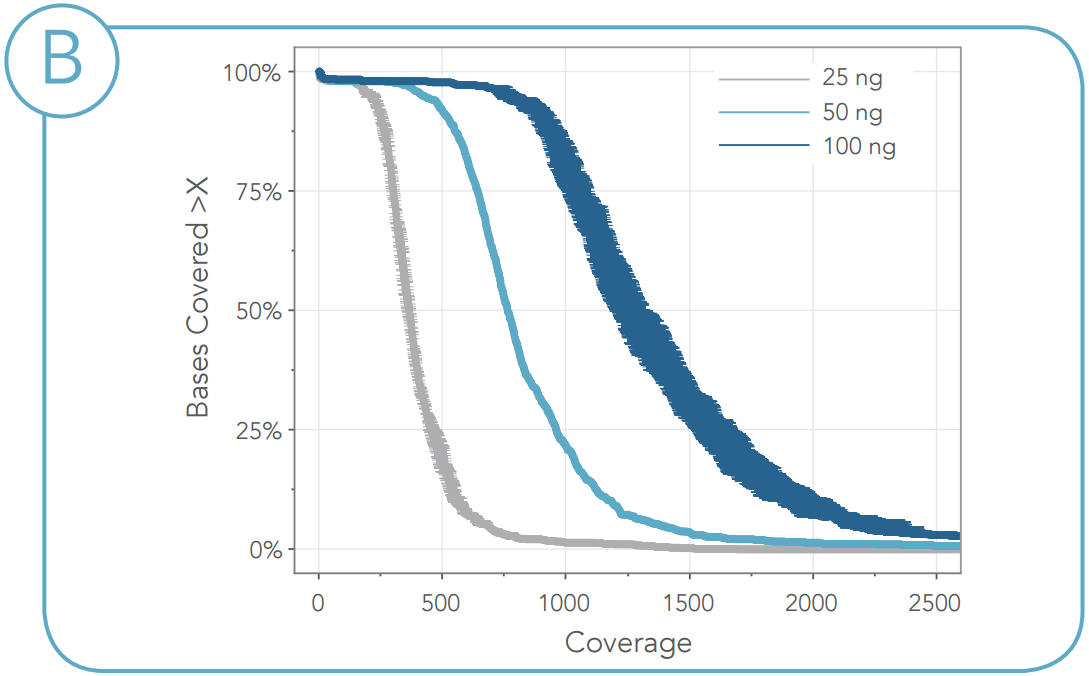 図6. FFPEサンプルの重複除去後のカバレッジ (A)インプット量が25 ng、50 ng、100 ngの場合について、重複除去後のカバレッジの平均値を示しています。エラーバーは反復測定値の標準偏差を表しています(n=3)。 (B)サンプル入力量によるカバレッジの平均値を示しています。x軸のエラーバーはカバレッジの標準偏差を表しています。 |
セルフリーDNAによるカバレッジの結果
結論
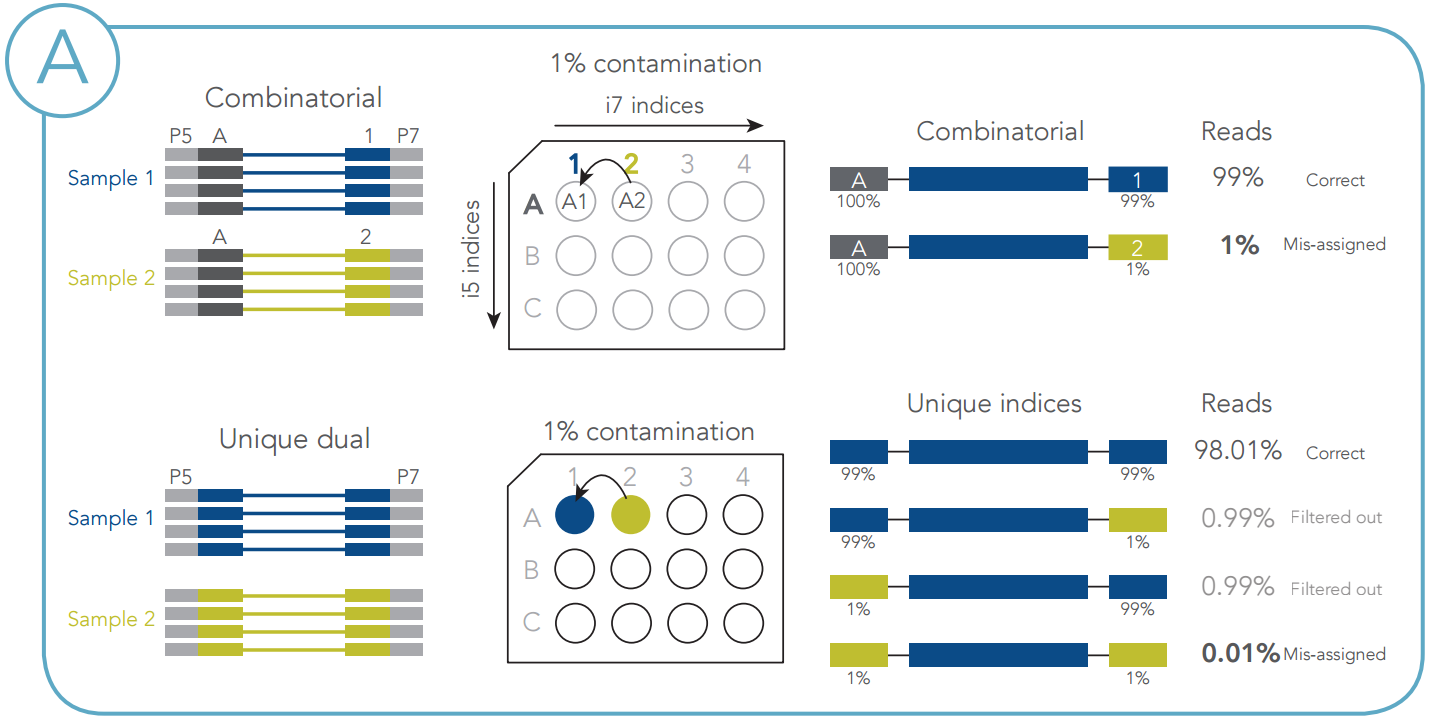
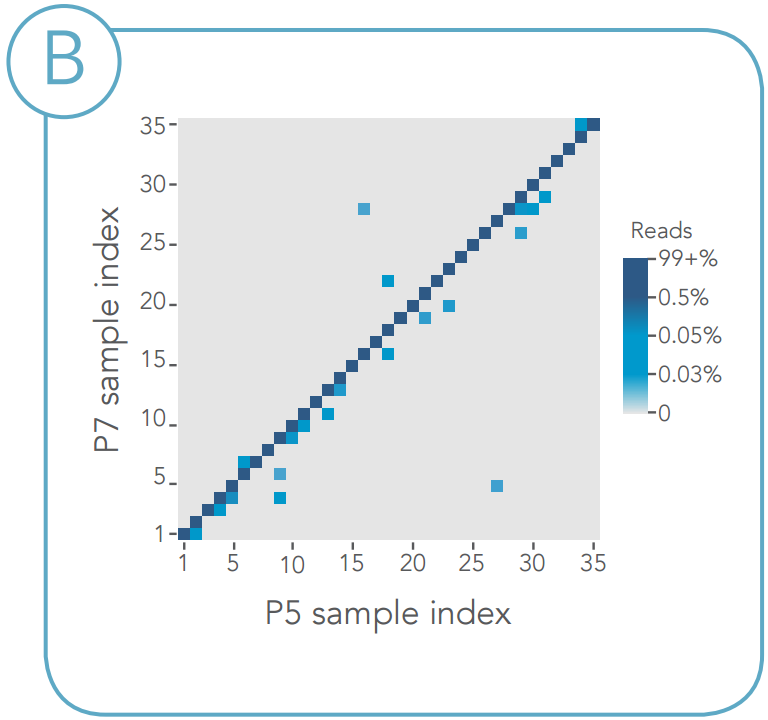
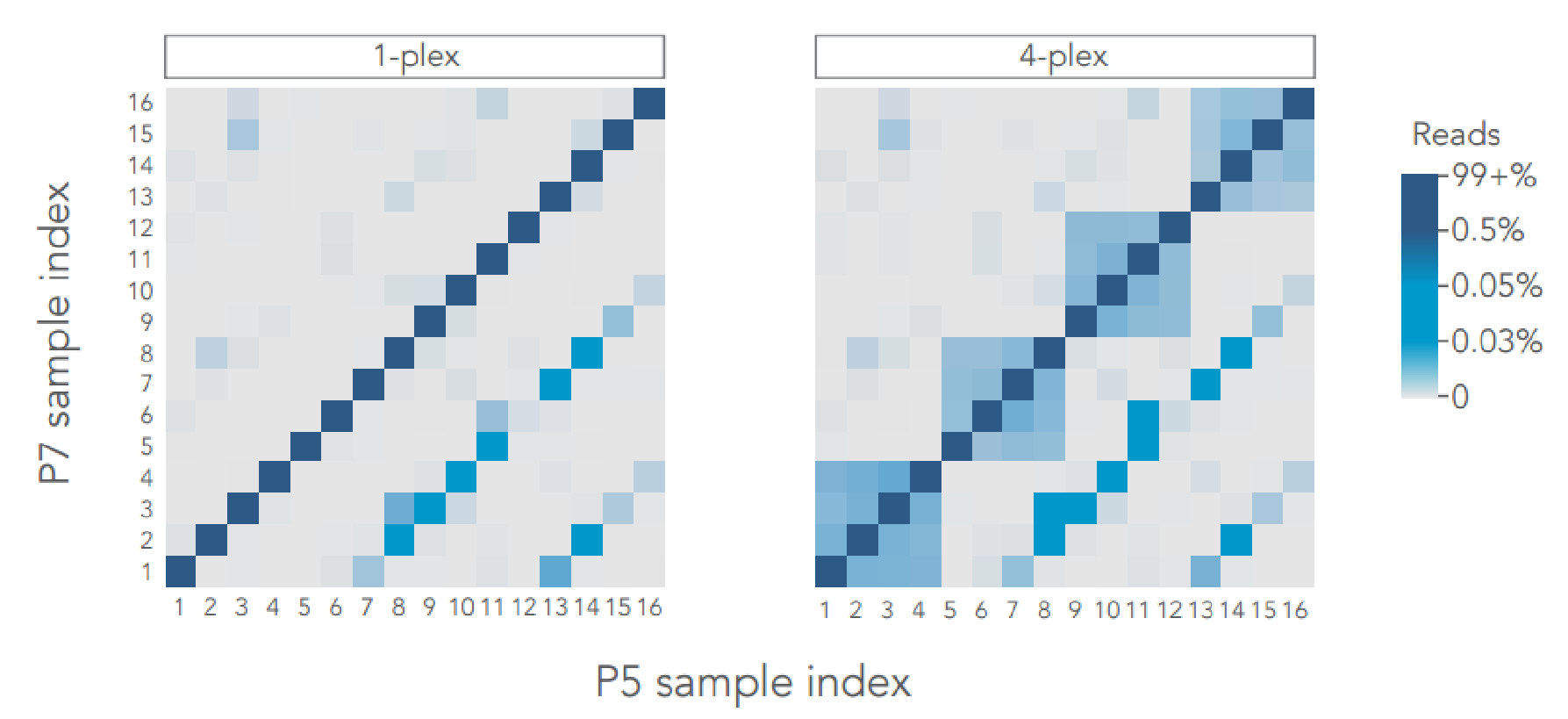
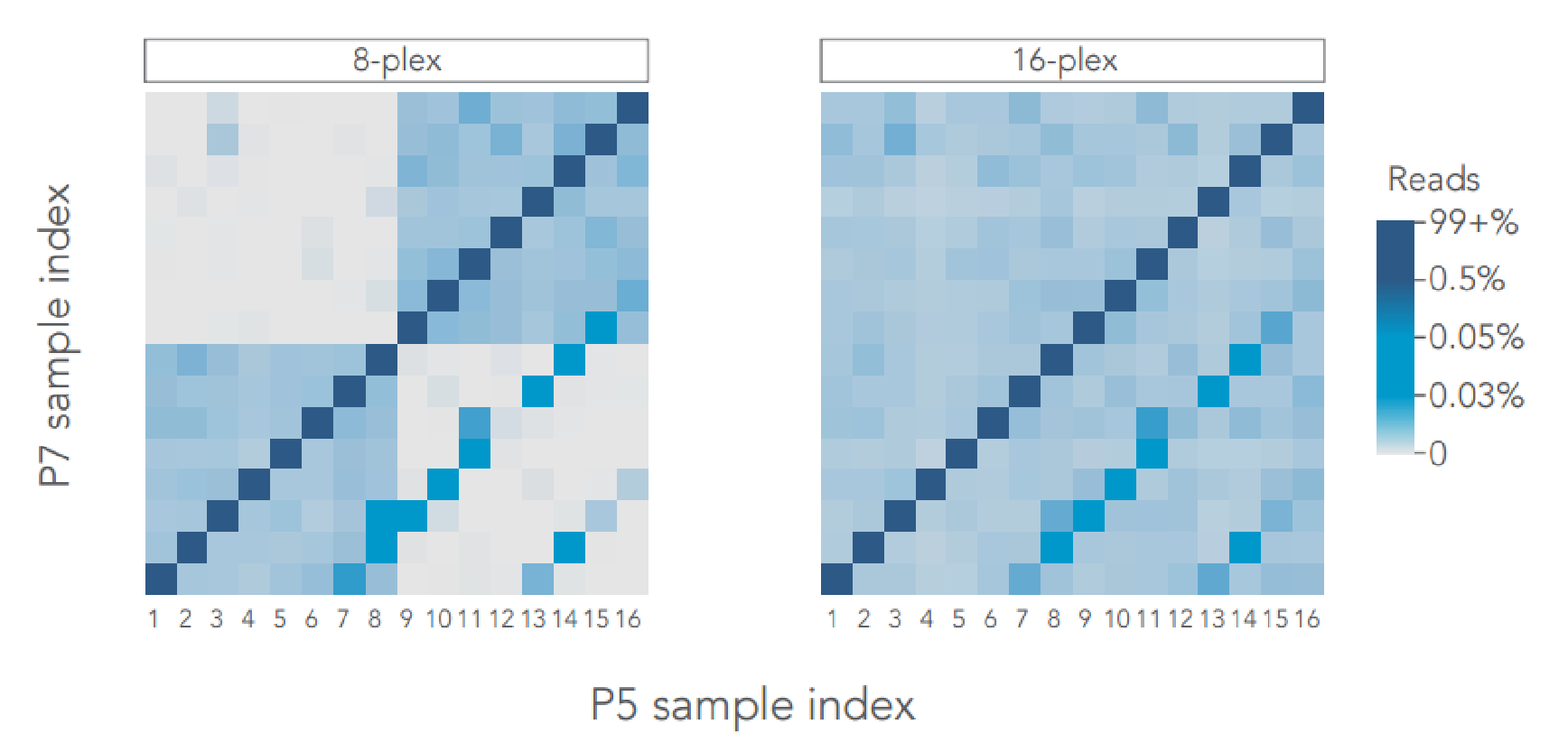
■ ユニークなデュアルインデックスアダプター配列は、マルチプレックスを用いる研究では、低頻度変異を正確に割り当てるのに欠かせません。この配列は、バーコードコンタミネーションやインデックスホッピングをより正確に除去できます。
■ コンセンサス配列を構築すると、in silico でエラーを修正でき、変異コールの特異度が劇的に向上します。
■ デュアルインデックスとUMIによるエラー修正の併用は、FFPEやセルフリーDNAなど、少量のインプット量しか出来ないサンプルや、扱いが難しいサンプルに対して非常に有用です。
■ コンセンサス配列を構築すると、in silico でエラーを修正でき、変異コールの特異度が劇的に向上します。
■ デュアルインデックスとUMIによるエラー修正の併用は、FFPEやセルフリーDNAなど、少量のインプット量しか出来ないサンプルや、扱いが難しいサンプルに対して非常に有用です。
References
原文: xGen® Dual Index UMI Adapters
resolve index hopping and enable
low-frequency variant detection
翻訳・監修:浜本 雄次
翻訳・監修:浜本 雄次
製品フォーカス
xGen® 製品の特徴
xGen® 製品は、高品質なプローブを1本ずつ合成し、全てのプローブに品質管理及び納品量の標準化を行っています。SNPs、インデル、CNV、LOH、および転移の高感度の検出に有用で、既存のパネルを拡張したり、完全にカスタマイズされたパネルを作成する柔軟性も持ち合わせています。GC含量の多い領域が取得しにくいのはプローブの合成が難しいためですが、IDTでは、プローブ1本ずつ全てにおいて品質管理を行っているため、納品したプローブプール内に各プローブが確実に必要量含まれています。そのため、GCリッチな領域を含むターゲット領域全体を均一に取得可能です。
» xGen®製品の特徴について詳しくはこちらをご覧下さい。
診断用のxGen Lockdownプローブも利用できます。 詳細は、japan-cc@idtdna.comにお問い合わせください。
xGen® Lockdown® Probes
新規プローブセットの作製や、お手持ちのプローブセットの拡張・補填など実験目的に合わせたプローブセットをご購入いただけます。各遺伝子から目的の遺伝子をお選びいただくプレデザイン品と、ご希望のプローブ配列を合成するカスタム品をご用意しております。» xGen® Lockdown® Probesの詳細についてはこちらをご覧ください。
xGen® Lockdown® Panels
- エクソームおよび各疾患用に最適化されたプローブセットです。
- ・ xGen® Exome Research Panel - エクソームシーケンス用プローブセット
- ・ xGen® Acute Myeloid Leukemia Cancer Panel - AML(急性骨髄性白血病)用プローブセット
- ・ xGen® Pan-Cancer Panel - 12種のがんに共通して変異が見られる127遺伝子のプローブセット
- ・ xGen® Inherited Diseases Panel - HGMD®(Human Gene Mutation Database)に基づいた遺伝性疾患に関わる領域及びSNPsのプローブセット
- ・ xGen® Human ID Research Panel - ヒトDNAの識別用に設計されたプローブセット
- ・ xGen® Human mtDNA Research Panel - ヒトミトコンドリアDNAを網羅的に解析可能なプローブセット
- ・ xGen® CNV Backbone Panel—Tech Access - ヒトゲノム全体のCNV(コピー数多型)を解析するプローブセット
» xGen® Lockdown® Panelsの詳細については、こちらをご覧ください。
xGen® Universal Blocking Oligos
アダプター配列同士の結合を防ぐブロッキングオリゴです。xGen® Universal Blockers – TS MixはIllumina社のインデックスアダプター用に設計されており、シングルインデックスとデュアルインデックス用のどちらのタイプにもready-to-useで使用可能です。3’末端のC3スペーサー付加など非特異な配列の結合・増幅を防ぐ修飾がされており、オンターゲット率を大幅に向上させます。» xGen® Universal Blockers – TS Mixの詳細については、こちらをご覧ください。
Additional reading
NGSターゲットキャプチャーの成功度を高めるプロトコールのヒント
プローブキャプチャー法よるエンリッチメント実験を行う際に、これらに注意することでより良いデータがで得られる可能性のある事柄を列挙しました。FFPEサンプルのDNAエンリッチメント時に、確実なシーケンスデータを得るための推奨事項
FFFEサンプルに対してエンリッチメントを行う際に、品質によってサンプル量を変えなければ、解析に十分なデータが得られません。こちらのアプリケーションノートでは、どの程度の品質の時に、どのようなデータが得られるのかを実際の実験データを用いて紹介しています。プローブキャプチャーによるマルチプレックスに必要なサンプル量とデータの品質
プローブキャプチャー法でマルチプレックスを行った際に、一度にマルチプレックスするサンプル数によるデータの品質がどのように影響するか、を実際の実験データを用いて紹介しています。また、サンプル量により、得られるデータの品質がどのように変わるかも併せて紹介しています。