NGSターゲットキャプチャーの成功度を高めるプロトコールのヒント
ライブラリーのエンリッチメントと最大パフォーマンス達成の手引き
ターゲットキャプチャーの実験における原理を正確に理解すると、解析によって得られるデータから、より多くの事象を明確化できるようになります。
進化する次世代シーケンシング(NGS)のワークフロー(図1)とIDTが提供するターゲットエンリッチメント製品群を補完するために、xGenハイブリダイゼーションキャプチャープロトコール(xGen hybridization capture of DNA libraries)を用いて信頼性と再現性が高い結果を得るための具体的なガイドラインを作成しました。
進化する次世代シーケンシング(NGS)のワークフロー(図1)とIDTが提供するターゲットエンリッチメント製品群を補完するために、xGenハイブリダイゼーションキャプチャープロトコール(xGen hybridization capture of DNA libraries)を用いて信頼性と再現性が高い結果を得るための具体的なガイドラインを作成しました。
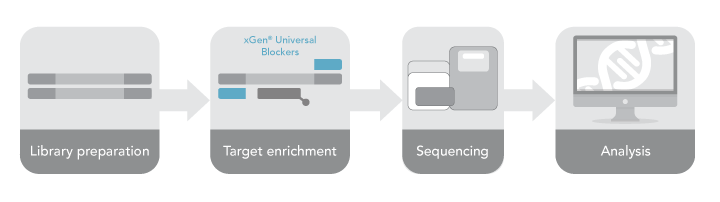 図1. NGSワークフローの概要 NGSワークフローは、DNAライブラリーの調製、ターゲットエンリッチメント、各プラットフォームによるサンプルのシーケンス、参照ゲノムに対してリードをコンピュータ計算でマッピングするデータ解析からなっています。 |
NGSターゲットキャプチャーの性能向上のヒント
- xGen NGS関連製品を使用して最高の結果を出す
xGen NGS関連製品には、xGen Lockdown Probes、 xGen Lockdown Panels、xGen Blocking Oligos、 xGen Dual Index UMI Adapters-Tech Access、 xGen Hybridization、Wash Kit、 xGen Library Amplification Primer Mixなどがあり、これらを用いることで最高の結果を出せます。
カスタムプローブの場合はIDT Target Capture Probe Design & Ordering Toolをご利用ください。(IDT 日本法人のカスタマーケアにご依頼頂いても構いません。(Japan Customer Care))xGenハイブリダイゼーションキャプチャーシステムは、確実で再現性の高いNGSの結果データを出すことに特化して最適化されています。
- お使いのNexteraやTruSeqというライブラリータイプに適したxGen Blocking Oligoを選ぶ
NexteraライブラリーとTruSeqライブラリーでは、対応するxGen Blocking Oligoが異なります。正しいxGen Blocking Oligoを用い、他のブロッキングオリゴに置き換えないことによって、キャプチャー効率全体を大幅に向上させることができます。 - キャプチャー実験のターゲットがヒトでない場合は、Human Cot DNAは最適ではない場合があることを理解する
最高の結果を出すには、マウスCot DNAやサケ精子DNAなどの代替品をお使いください。 - 実験で用いる実験装置が最近キャリブレートされたことを確認する
ハイブリダイゼーション反応温度や洗浄温度のわずかな変化(+/-2℃)もオンターゲット率とGCバイアスに影響します。
NGSデータのGCの偏りによるバイアスは、キャプチャー均一性の悪さの指標であることが知られているため、キャリブレーションは考慮すべき重要事項です。洗浄温度が高いと、低GC含量領域がキャプチャーされなくなってしまいます。洗浄温度が低いと、オンターゲット率が下がります。
注:ターゲットエンリッチメントシーケンスでは、シーケンシングリード数のうち、いくつがオンターゲットで、いくつがオフターゲットであるかを算出することが大切です。
最も単純な方法は、オンターゲット領域にあるアライメントされた塩基の数を計算することです。しかし、この方法は、さまざまなインサート長のライブラリーに適用しようとすると正しい比較ができない場合があります。例えば、キャプチャーしたいターゲットのうちのひとつが150 bpのリードであり、インサートが150 bpであれば、オンターゲット塩基は100%になります。
一方、もっと大きな300 bpのリードが同じ150 bpのターゲットにアライメントすると、塩基の50%だけがオンターゲットだということになります。分析を簡素化し、多様なサンプル間で比較を行うには、通常、ターゲット領域の両端もオンターゲット領域となるようにし(両側に150 bp)、フランキング領域を加えたオンターゲット塩基の計算を行います(図2)。

図2. フランキングされたオンターゲットのシーケンシング解析
多様なサンプルのデータ解析中に、オンターゲットとオフターゲットのシーケンシングリード数の測定法としてよく用いられるものに、ターゲット領域の両端もオンターゲット領域となるよう、フランキングされたオンターゲット塩基を計算する方法があります。
- 複数のサンプルを処理する場合は、プレートプロトコールを用いる
プレートプロトコールを用いると(チューブプロトコールよりも)1実験内のサンプル間の操作によるバラツキが小さくなります。 - プレートを使って作業する場合は、プレートの外縁のウェルは使用しない
外縁のウェルでは蒸発が起こりやすいからです。 - ハイブリダイゼーション反応チューブ/プレートが密閉されていることを必ず確認する
ハイブリダイゼーション反応で蒸発が起こるとキャプチャーの失敗を招くことがあります。ヒートシーラーを用いる場合は、シーリング方法の最適化が必要になることがあります。 - ハイブリダイゼーションのインキュベーション時間を4時間から16時間に延長することで、特にパネルが小さい(1000プローブ以下)場合に、より良い結果を取得できることがある。
- 65℃の処理が必要になるwash bufferを、使用前に15分以上予熱する
予熱すると、プロトコール中の重要ステップでbufferを65℃まで加熱するのに十分な時間の確保に役立ちます。 - オプションのAppendix A: AMPure XP Bead DNA concentration protocolを用いる場合は、誤ってSPRIビーズがハイブリダイゼーション反応にキャリーオーバーしていないことを確認する
ビーズのキャリーオーバーは、オンターゲット率とカバレッジに悪影響を及ぼします。 - オプションのAppendix A: AMPure XP Bead DNA concentration protocolは、標準的なSpeedVac法よりもHuman Cot DNAの必要量が多いことを理解する
追加のHuman Cot DNA(IDT Cat#1080768; 1080769)は、xGen Hybridization and Wash Kitとは別に購入する必要があります。AMPure XP Bead DNAを用いたプロトコールで標準量のHuman Cot DNAを用いると、AMPure XPによる濃縮中にヒトCot DNAフラグメント(フラグメントサイズ50~300 bp)が損失することによって、オンターゲット率の低下を招くことがあります。 - キャプチャーの反応速度を高めるため、45分間のビーズキャプチャー中、10~12分ごとにボルテックスを行う
- プロトコールの各ステップでStreptavidinビーズを乾燥させない
必要な場合は、洗浄時間をわずかに延ばして、ビーズを乾燥させないようにしてください。 - 室温でのwash中にStreptavidinビーズが完全に再懸濁状態であることを確認する
室温wash中に十分にボルテックスし、インキュベーションガイドラインを守って操作を行うと、データの質が向上します。注:プレートシール上にビーズが飛散してもキャプチャーの結果に悪影響はありません。 - プレートで接着シールを用いる必要があるプロトコールでは、各ステップで必ず新しいシールを用いる
新しいシールを使用すると、プレートプロトコールを実施している間、サンプルのクロスコンタミネーションを回避できます。
ゲノムDNAから調製したライブラリーのターゲットエンリッチメントのために従うべきガイドラインと必要なステップはIDT xGen hybridization capture of DNA libraries protocolに記載されています。
xGen Lockdown Probes/Panelsと併用すると最高の結果を期待できます。アダプター配列により起こる非特異的な結合を減らすには、xGen Universal Blockersを使用します。
これらの信頼性の高い製品とプロトコールガイドラインは、お客様のNGSターゲットエンリッチメントアプリケーションを成功に導きます。
xGENハイブリダイゼーションキャプチャー関連製品についてはxGen family of hybridization capture productsでさらに詳しくご覧ください。
References
原文:Protocol tips to boost your NGS target capture success
著者:Hana Johannesen, Scientific Writer, IDT.
翻訳・監修:浜本 雄次
著者:Hana Johannesen, Scientific Writer, IDT.
翻訳・監修:浜本 雄次
製品フォーカス
xGen® 製品の特徴
xGen® 製品は、高品質なプローブを1本ずつ合成し、全てのプローブに品質管理及び納品量の標準化を行っています。SNPs、インデル、CNV、LOH、および転移の高感度の検出に有用で、既存のパネルを拡張したり、完全にカスタマイズされたパネルを作成する柔軟性も持ち合わせています。GC含量の多い領域が取得しにくいのはプローブの合成が難しいためですが、IDTでは、プローブ1本ずつ全てにおいて品質管理を行っているため、納品したプローブプール内に各プローブが確実に必要量含まれています。そのため、GCリッチな領域を含むターゲット領域全体を均一に取得可能です。
» xGen®製品の特徴について詳しくはこちらをご覧下さい。
診断用のxGen Lockdownプローブも利用できます。 詳細は、japan-cc@idtdna.comにお問い合わせください。
xGen® Lockdown® Probe Pools
新規プローブセットの作製や、お手持ちのプローブセットの拡張・補填など実験目的に合わせたプローブセットをご購入いただけます。各遺伝子から目的の遺伝子をお選びいただくプレデザイン品と、ご希望のプローブ配列を合成するカスタム品をご用意しております。» xGen® Lockdown® Probesの詳細についてはこちらをご覧ください。
xGen® Lockdown® Panels
- エクソームおよび各疾患用に最適化されたプローブセットです。
- ・ xGen® Exome Research Panel - エクソームシーケンス用プローブセット
- ・ xGen® Acute Myeloid Leukemia Cancer Panel - AML(急性骨髄性白血病)用プローブセット
- ・ xGen® Pan-Cancer Panel - 12種のがんに共通して変異が見られる127遺伝子のプローブセット
- ・ xGen® Inherited Diseases Panel - HGMD®(Human Gene Mutation Database)に基づいた遺伝性疾患に関わる領域及びSNPsのプローブセット
- ・ xGen® Human ID Research Panel - ヒトDNAの識別用に設計されたプローブセット
- ・ xGen® Human mtDNA Research Panel - ヒトミトコンドリアDNAを網羅的に解析可能なプローブセット
- ・ xGen® CNV Backbone Panel—Tech Access - ヒトゲノム全体のCNV(コピー数多型)を解析するプローブセット
» xGen® Lockdown® Panelsの詳細については、こちらをご覧ください。
xGen® Universal Blocking Oligos
アダプター配列同士の結合を防ぐブロッキングオリゴです。xGen® Universal Blockers – TS MixはIllumina社のインデックスアダプター用に設計されており、シングルインデックスとデュアルインデックス用のどちらのタイプにもready-to-useで使用可能です。3’末端のC3スペーサー付加など非特異な配列の結合・増幅を防ぐ修飾がされており、オンターゲット率を大幅に向上させます。» xGen® Universal Blockers – TS Mixの詳細については、こちらをご覧ください。
Additional reading
xGen® Dual Index UMI Adaptersによるインデックスホッピングの解消と低頻度変異の検出
インデックスホッピングや低頻度変異の検出に有用なDual Index Apadtersについての説明と、このアダプターを用いて得られた具体的なで実験データを開示しています。プローブキャプチャーによるマルチプレックスに必要なサンプル量とデータの品質
プローブキャプチャー法でマルチプレックスを行った際に、一度にマルチプレックスするサンプル数によるデータの品質がどのように影響するか、を実際の実験データを用いて紹介しています。また、サンプル量により、得られるデータの品質がどのように変わるかも併せて紹介しています。