Affinity Plus qPCR Probes
Affinity Plus qPCR プローブは、FFPE組織やリキッドバイオプシーなどの、検出が難しいターゲットや、トランスクリプトバリアントなどのSNPジェノタイピングに有益です。
Affinity Plus qPCR プローブは最大6個までの Locked Nucleic Acid (LNA)塩基を含みます。LNAはDNA塩基と比較して安定であるため、プローブにこのLNA塩基を用いると、ハイブリダイゼーション時のTm値が向上します。
LNAを用いることで、下記が期待できます。
・一般的なqPCRと比較して、ミスマッチの識別能が向上します。
・アッセイにおけるハイブリダイゼーションの特異性を高める簡単なアプローチです。
・MGB修飾よりも簡単にTm値を調整できます。
LNAについては下記の記事でも詳しく紹介しています。
Improve assays with customizable oligos and probes containing cost-effective locked nucleic acids
Affinity Plus qPCR プローブは最大6個までの Locked Nucleic Acid (LNA)塩基を含みます。LNAはDNA塩基と比較して安定であるため、プローブにこのLNA塩基を用いると、ハイブリダイゼーション時のTm値が向上します。
LNAを用いることで、下記が期待できます。
・一般的なqPCRと比較して、ミスマッチの識別能が向上します。
・アッセイにおけるハイブリダイゼーションの特異性を高める簡単なアプローチです。
・MGB修飾よりも簡単にTm値を調整できます。
LNAについては下記の記事でも詳しく紹介しています。
Improve assays with customizable oligos and probes containing cost-effective locked nucleic acids
Affinity Plus qPCR Probesの特長
qPCRプローブの特異性を高めるために、Affinity PlusなどのLocked Nucleic Acid (LNA) 塩基が有用です[1-4]。LNA塩基はTmを大幅に増加させるため、Affinity Plus qPCR プローブは標準的なプローブと比較して、プローブ長を短くできます。プローブが短くなるほど、消光性が高くなり、S/N比も高くなります。
さらに、Affinity Plus qPCR Probesは、変異や一塩基多型(SNP)を判別することができます[1]。Affinity Plus qPCR probesには、LNA塩基を複数個デザインすることができ、15℃以上のΔTmが得られます。これにより、リアルタイムPCRや、多型を見分けるためのハイブリダイゼーション法などでの精度が飛躍的に向上します。
さらに、Affinity Plus qPCR Probesは、変異や一塩基多型(SNP)を判別することができます[1]。Affinity Plus qPCR probesには、LNA塩基を複数個デザインすることができ、15℃以上のΔTmが得られます。これにより、リアルタイムPCRや、多型を見分けるためのハイブリダイゼーション法などでの精度が飛躍的に向上します。
Affinity Plus qPCR Probes mini スケール
Affinity Plus qPCR Probeをお試し頂きやすい小さなスケールで提供しております。
FAM、HEX、Yakima Yellow(YAK)の3種類の蛍光色素と、Iowa Blackクエンチャーの組み合わせでご利用いただけます。
FAM、HEX、Yakima Yellow(YAK)の3種類の蛍光色素と、Iowa Blackクエンチャーの組み合わせでご利用いただけます。
| 蛍光色素 | クエンチャー | 納品量 | 価格 | 納期 |
|---|---|---|---|---|
| 5' 6-FAM | 3' Iowa Black RQ-Sp | 0.5 nmol | ¥ 9,900 | 約2週間 |
| 5' HEX | 3' Iowa Black RQ-Sp | 0.5 nmol | ¥ 9,900 | |
| 5' Yakima Yellow | 3' Iowa Black RQ-Sp | 0.5 nmol | ¥ 9,900 |
Affinity Plus qPCR Probes 価格
| 蛍光色素 | クエンチャー | 合成スケール | |
|---|---|---|---|
| 250 nmole | 1 µmole | ||
| 5' 6-FAM | 3' Iowa Black RQ-Sp | ¥ 30,900 | ¥ 43,100 |
| 3' Black Hole Quencher 1 | ¥ 37,000 | ¥ 53,100 | |
| 5' HEX | 3' Iowa Black RQ-Sp | ¥ 32,600 | ¥ 45,300 |
| 3' Black Hole Quencher 1 | ¥ 38,700 | ¥ 54,700 | |
| 5' Yakima Yellow | 3' Iowa Black RQ-Sp | ¥ 32,600 | ¥ 45,300 |
| 5' TYE 665 | 3' Iowa Black RQ-Sp | ¥ 47,000 | ¥ 66,300 |
| 3' Black Hole Quencher 2 | ¥ 52,000 | ¥ 73,000 | |
| 5' TYE 563 | 3' Iowa Black RQ-Sp | ¥ 47,000 | ¥ 66,300 |
| 3' Black Hole Quencher 2 | ¥ 52,500 | ¥ 73,000 | |
| 5' TEX 615 | 3' Iowa Black RQ-Sp | ¥ 35,900 | ¥ 49,200 |
| 3' Black Hole Quencher 2 | ¥ 54,200 | ¥ 75,700 | |
| 5' Cy5 | 3' Iowa Black RQ-Sp | ¥ 35,400 | ¥ 49,200 |
| 3' Black Hole Quencher 2 | ¥ 42,000 | ¥ 59,700 | |
| 5' Cy3 | 3' Iowa Black RQ-Sp | ¥ 34,300 | ¥ 48,100 |
| 3' Black Hole Quencher 2 | ¥ 41,400 | ¥ 58,600 | |
Affinity Plus qPCR Probes デザイン方法
Affinity Plus LNA塩基の導入によるTm値
配列に応じてLNA塩基を挿入することにより、Tmが3 ~ 6℃上昇します。LNAを複数個追加することで、ΔTmが15℃を超すAffinity Plus qPCRプローブをデザインできます。
Affinity Plus LNA塩基のTmは、Affinity Plus:Affinity Plus > Affinity Plus : DNA > DNA : DNA です。
そのため、セルフダイマーとヘアピンを確認し、Affinity Plus : Affinity Plus のペアリングを最小限にする事が大切です。
OligoAnalyzerを用いることで、これらの二次構造を確認できます。
SNP解析における Affinity Plus LNA塩基の推奨個数と配置箇所
・Affinity Plus qPCR Probeでは、最大で6個のAffinity Plus LNA塩基の使用をお勧めしています。・SNP解析にAffinity Plus qPCR Probesを使用する際は、Affinity Plus LNA塩基は、「SNPサイト」と「SNPと隣接する塩基」に配置する必要があります。
その際、可能であればSNPはプローブの中心に配置して下さい。
・Affinity Plus LNA塩基をプローブの端に設定するのは避けてください。
・Affinity Plus LNA塩基の連続は4つ以下にしてください。
Affinity Plus LNA塩基を導入する配列とTm値の関係
Affinity Plus LNA塩基の配置はその配列に依存します。そのため、SNP解析用プローブがSNPに対してミスマッチする場合と、マッチする場合のΔTmを十分な値にするためには、配列の最適化を行う必要がります。qPCRの場合、アンプリコンサイズは150 bpまでを推奨します。
ただし、FFPEや血中DNA(circulating DNA)などの断片化された配列の場合は、デジタルPCR (dPCR)や、
80 ~ 100 bpの短いアンプリコンを推奨します。
IDTにデザインを依頼する
IDTにデザインを依頼する場合は、下記のお問い合わせフォームからご依頼ください。| Affinity Plus qPCR Probes デザイン依頼 | |
|---|---|
| To:japan-cc@idtdna.com | |
Affinity Plus qPCR Probes を用いて取得したデータ例
■ 安定性の向上
Affinity Plus LNA 塩基を含む配列は、他社のLNA配列と同等のアニーリングプロパティを提供します(図1)。 ハイブリダイゼーションの親和性の向上により、Tm値も増加し、in vitro および in vivoの両方で、配列の安定性が向上します。
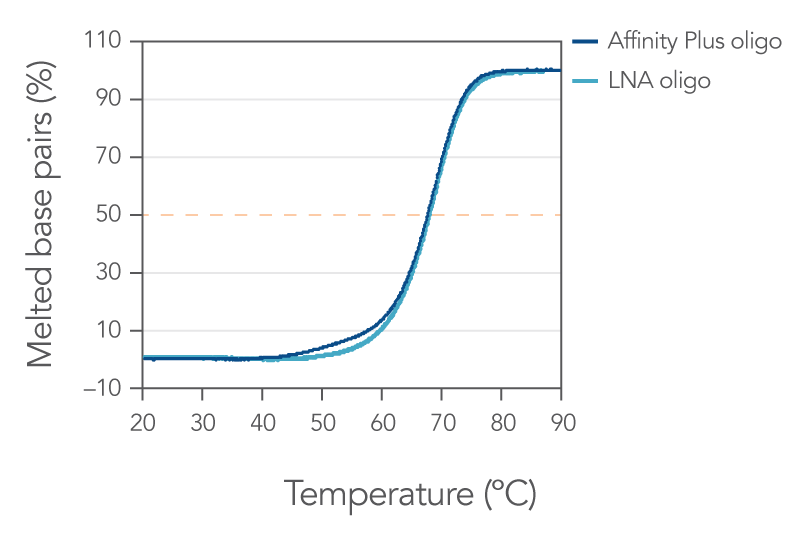 図1. Affifinity Plus Locked nucleic acidsを含む二本鎖DNAは、他社で合成されたLNAと同様のアニーリング特性を示す。 15塩基の配列 [GGTCCT+T+A+CTTGGTG]は、+T、+A および +C部位に Affinity Plus もしくは LNA 修飾を組み込んで合成されました。 これらのオリゴは、1 M Na+ バッファー (pH 7) で各配列の相補鎖と混ぜ合わされ、 Owczarzy, et al., Biochemistry (2004), 43:3537–554を基にメルティングカーブを描画しました。 乖離した塩基対の割合は、温度の関数としてプロットされています。本実験は7回以上の冷却および過熱により得られています。 融解温度は、Affinity Plus オリゴで67.7 ± 0.3℃、LNAオリゴで67.9±0.3ºC であると測定されました。 37℃での二本鎖ハイブリダイゼーションの自由エネルギーを計測したところ、Affinity Plus オリゴでは、 –18.1±0.9 kcal/mol、LNAオリゴでは、 –18.7±0.9 kcal/mol でした。 これらの結果から、AffinityPlus および LNA 修飾におけるLocked nucleic acidは、同等のアニーリング特性を有します。
|
■ LNA塩基を配置することによるTm値の変化
未修飾のプローブ配列と比較して、LNAを含んだAffinity qPCRプローブは高いTm値を示し、特にGC含量の低い領域において優れた安定性を示します。安定性が向上するため、ターゲット領域のサイズが限られている場合でも、より短いプローブのデザインも可能です。図2は、Affinity Plus qPCR プローブが、PrimeTime qPCR LNAプローブと同等のプロファイルを提供することを示しています。両プローブとも、未修飾のプローブと比較して、より迅速なターゲットの増幅を示しています。
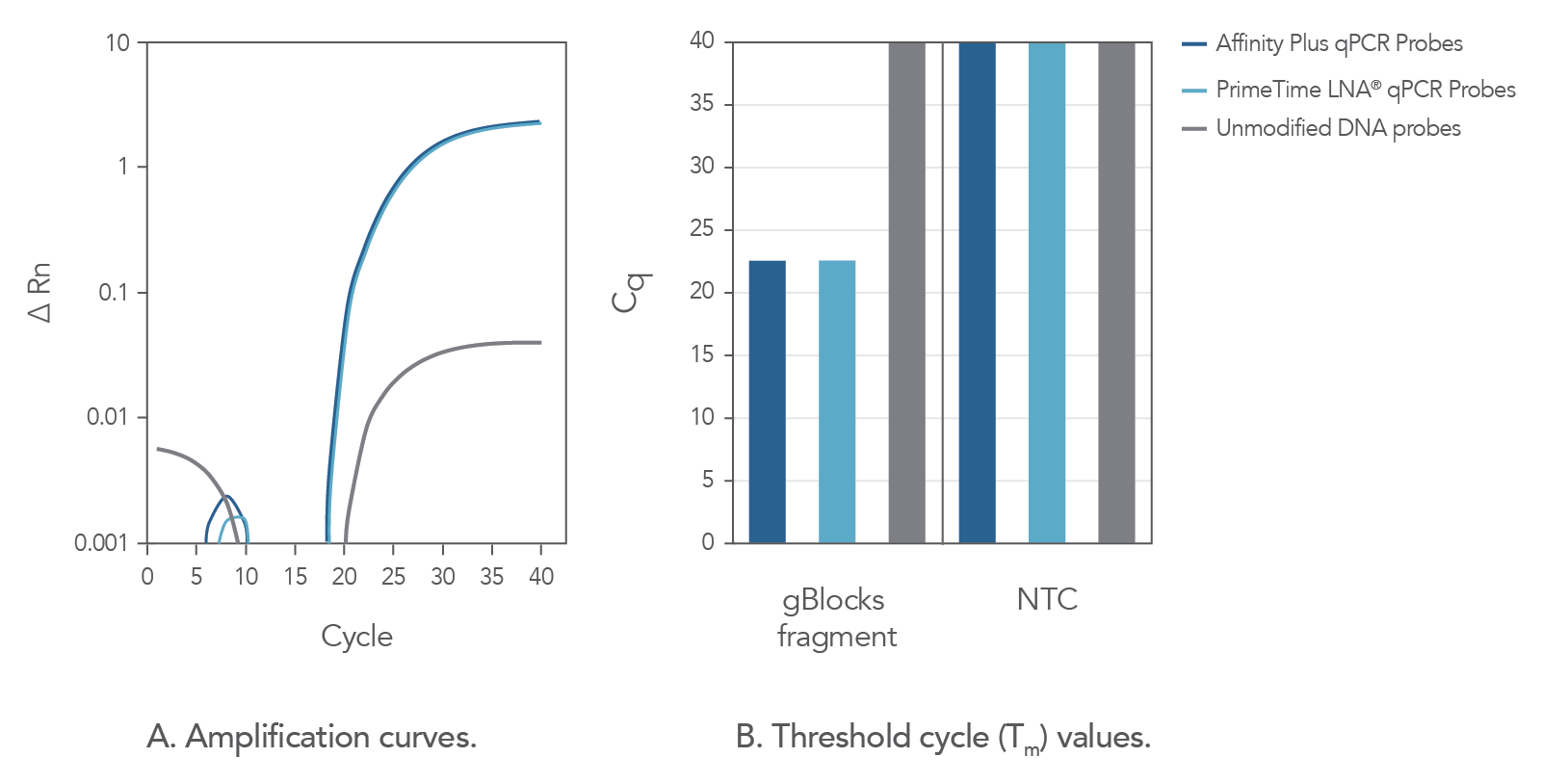 図2. Affinity Plus qPCRプローブと、PrimeTime LNA qPCRプローブは同等の実験結果を示す。 RSウイルスのA株から、M2-2遺伝子を検出するために、カスタムでPrimeTimeを用いてqPCRアッセイを設計しました。 gBlocks Gene Fragmentsをターゲットテンプレートとして用い、PrimeTime LNA qPCR プローブ、Affinity Plus qPCR プローブを、Locked Nucleaic Acidを用いないDNA配列と比較しました。 PrimrTime LNA qPCR と、Affinity Plus qPCR プローブは、同じ位置に7つのLocked Nucleic Acidを配置し、Tm値 は 68.1℃でした。 対して、Locked Nucleic Acidを配置しないDNA配列のTm値は49.9℃でした。 テンプレートとプローブのシーケンスを下記に示し、Locked Nucleic Acidの位置を[+N]で示します。 (A) 3種のプローブを使用した増幅曲線を描きました。Affinity Plus qPCR プローブ とLNA プローブでは、同じ結果をもたらす事を示しています。 (B)テンプレート(gBlocks)の有無による Cq値の比較 Locked Nucleic Acidを含む2種のプローブは、テンプレートを特異的に増幅することができました。 NTC = no template control. (テンプレートなし)
|
■ SNP解析
図3の4つのジェノタイピングアッセイのクラスタープロットは、LNAを含むAffinity Plus qPCRプローブが、PrimeTime LNA プローブと同等のコールであることを示しています(図3)。
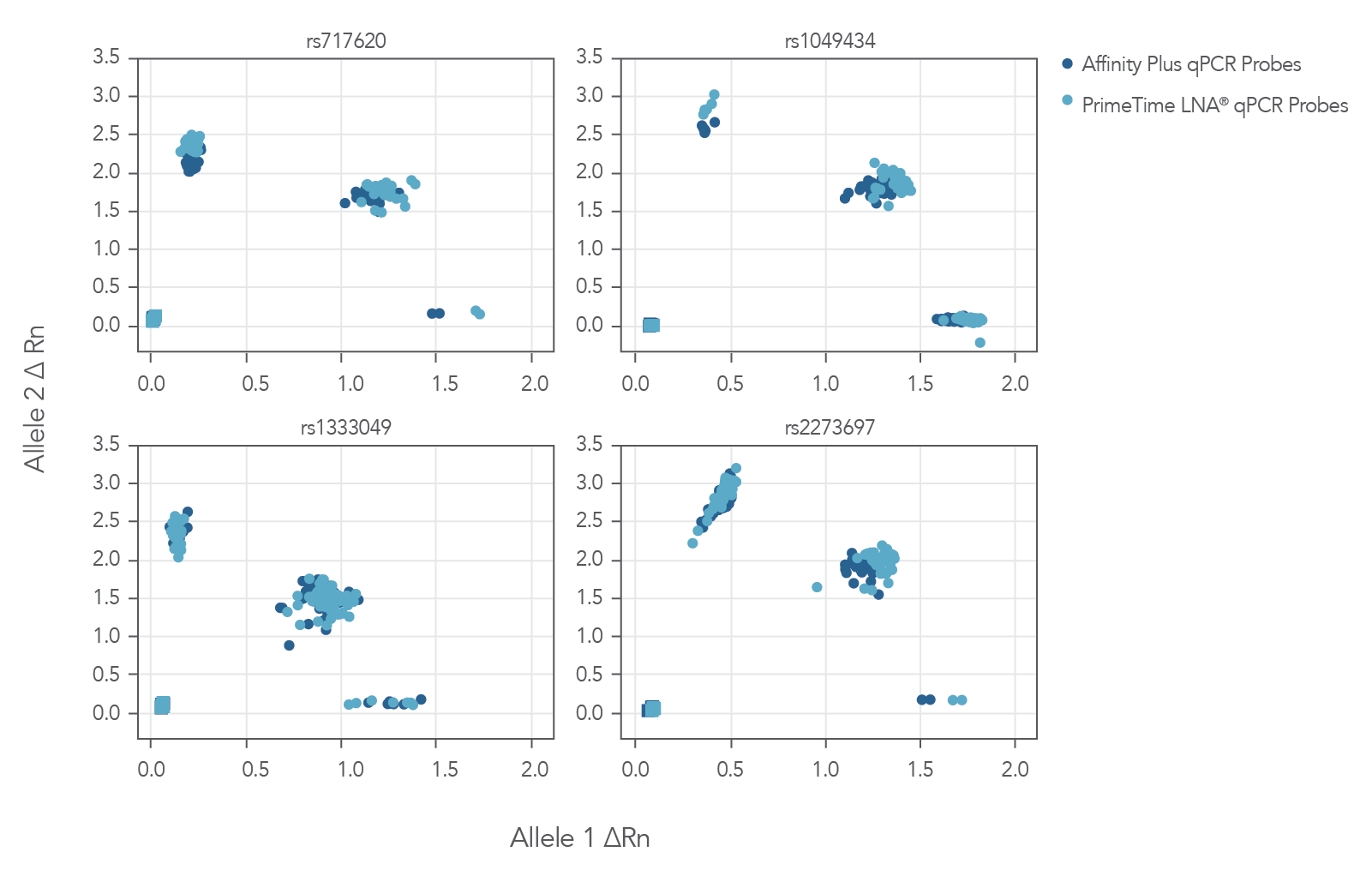 図3. Affinity Plus qPCR Probesは、PrimeTime LNA Probes と同等のコールを示す。 46種の異なるDNAサンプル(Coriell) に対し、同配列をAffinity Plus qPCR プローブ、PrimeTime LNA qPCR プローブとして合成したプローブを用いて4種のジェノタイピングアッセイを行いました。 Affinity Plus プローブとLNAプローブから得られた結果に明確な違いは見られませんでした。 得られたすべての解析結果は以前得られた結果と一致しました(データ非掲載)。 プライマーとプローブの配列は、Affinity Plus qPCR プローブのウェブページのパフォーマンスタブの表1に掲載しています。 rs1333049以外はすべてADME※アッセイです。 ※ADME (Absorption, Distribution, Metabolism and Excretion) アッセイで使用されるAffinity PlusプローブとPrimeTime LNAプローブの配列を以下に示します。
|
References
- Davialieva K, Kiprijanovska S, Plaseska-Karanfilska D. (2013) Fast, reliable and low cost user-developed protocol for detection, quantification and genotyping of hepatitis C virus. J Virol Methods, 196:104–112.
- Owczarzy R, You Y, et al. (2011) Stability and mismatch of locked nucleic acid–DNA duplexes. Biochemistry, 50(43):9352–9367.
- You Y, Moreira BG, et al. (2006) Design of LNA probes that improve mismatch discrimination. Nucl Acid Res 34(8):e60, doi:10.1093/nar/gkl175.
- Johnson MP, Haupt LM, Griffiths LR. (2004) Locked nucleic acid (LNA) single nucleotide polymorphism (SNP) genotype analysis and validation using real-time PCR. Nucleic Acids Res, 32(6):e55.